library(sp)
library(sf)
library(terra)
data(meuse)
data(meuse.grid)
library(tidyverse)
library(gstat)
library(GeoModels)Comparaison de l’efficatité de R et Julia en Geostatistiques
L’objectif est de comparer des packages R, et Julia pour du krigeage.
On va utiliser :
- R : gstat
- R : GeoModels
- Julia : GeoStats
On compare les points suivant :
- simplicité des données d’entrée
- concision du code
- vitesse d’exécution
- type d’estimation
Pour chaque package, on calculera pour les mêmes données le variogramme empirique, puis on ajustera un variogramme théorique et on fera un krigeage.
Données
On utilise les données meuse du package sp. Celles-ci contiennent des données de concentration en métaux près de la meuse, avec des coordonnées cartésiennes en m.
Pour travailler sur ces concentrations, on passera tout au log afin de les normaliser.
Pour la visualisation en R, on utilise le package terra.
0 - Le package terra
dernière version : 1.7-39 du 22/06/2023
Travail des données spatiales sous 2 formes : raster et vector.
raster = grilles rectangulaires, adapté aux données continues -> Classe SpatRaster
vector = points, lignes, polygones = données discrètes. -> classe SpatVector
Début de la doc = récap des méthodes importantes (il faut bien, la doc fait 300 pages)
Sert à manipuler, visualiser des objets spatiaux, notamment après les calculs pour simplifier le travail sur les résultats.
Méthode rast = créer un objet de classe SpatRaster. Le type de la 1ere entrée peut varier.
meuseRast = rast(meuse.grid,type="xyz",crs = "EPSG:2154")
meuseVect = vect(meuse,geom=(c('x','y')))meuseRastCd = rast(select(meuse,x,y,zinc),type="xyz")
log(meuseRastCd)class : SpatRaster
dimensions : 3898, 2786, 1 (nrow, ncol, nlyr)
resolution : 1, 1 (x, y)
extent : 178604.5, 181390.5, 329713.5, 333611.5 (xmin, xmax, ymin, ymax)
coord. ref. :
source(s) : memory
name : zinc
min value : 4.727388
max value : 7.516977 meuseRastCu = rast(select(meuse,x,y,copper),type="xyz")
meuseRastCuCd = c(meuseRastCd,meuseRastCd) #methode c() = combinaison de layers1 - Package R gstat
gstat est disponible sur le CRAN : https://CRAN.R-project.org/package=gstat
On peut y trouver également un tutoriel sur le dataset “meuse” utilisé ici, qui présentent le type de données spatiales utilisées par le package (spatial dataframe, données grillées) et les méthodes permettant de travailler avec (méthodes de visualisation du package sp, ajustement de variogrammes, directionnels ou non, krigeage, simulation conditionnelle).
La version la plus ancienne sur les archives du CRAN date de 2003.
Au départ, les données spatiales supportées étaient celles du package sp, mais les données de type sf le sont également.
INSERER AU BON ENDROIT LES CELLULES
Transformation au format sf
df = select(meuse,x,y,cadmium,zinc)
df_grid = select(meuse.grid,x,y)
df_sf <- df %>%
sf::st_as_sf( coords = c('x','y'))
df_sf <- sf::st_make_valid(df_sf)
df_grid_sf = df_grid%>% sf::st_as_sf(coords = c('x','y'))%>%sf::st_make_valid()
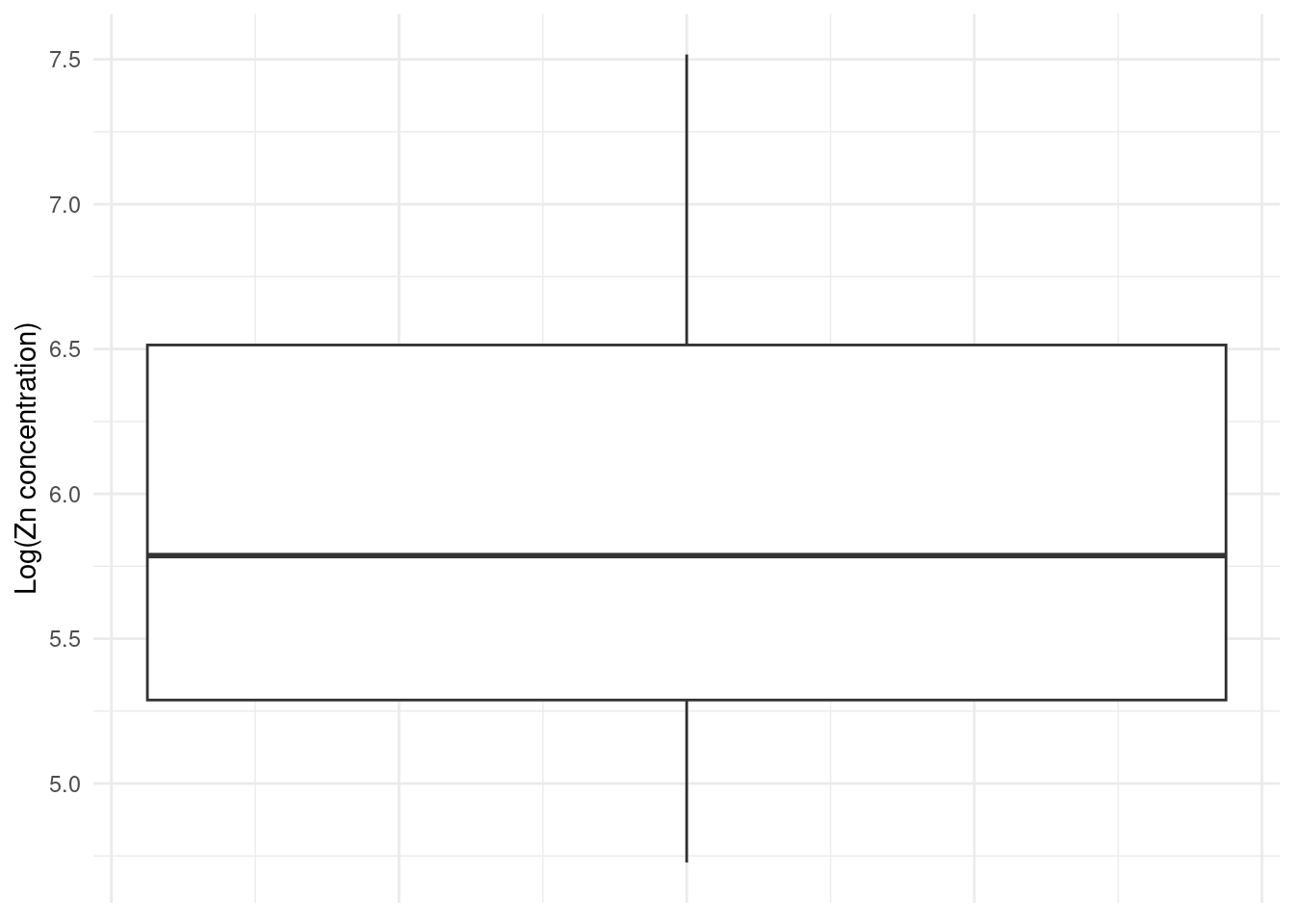
ggplot(df, aes(y=log(zinc))) +
geom_boxplot(notch=FALSE) +
labs(y = "Log(Zn concentration)") +
theme_minimal() +
theme(
axis.text.x = element_blank(),
axis.ticks = element_blank(),
axis.line = element_blank()
)
Variogramme empirique
Pour simplifier la comparaison avec le package Julia, on va se contenter d’une moyenne constante sans introduire de prédicteurs : on demande donc log(zinc)~1. Si on avait voulu, comme dans le tutoriel du package, expliquer la moyenne par la distance à la rivière, il faudrait modifier cette formule en log(zinc)~distance après avoir rajouté une colonne distance au sf dataframe.
Appeler plot sur un objet issu de variogram permet d’en obtenir un tracé.
start_time <- Sys.time()
test <- variogram(log(zinc)~1 , df_sf,cutoff = 2000)
end_time <- Sys.time()
end_time - start_timeTime difference of 0.02656889 secsplot(test)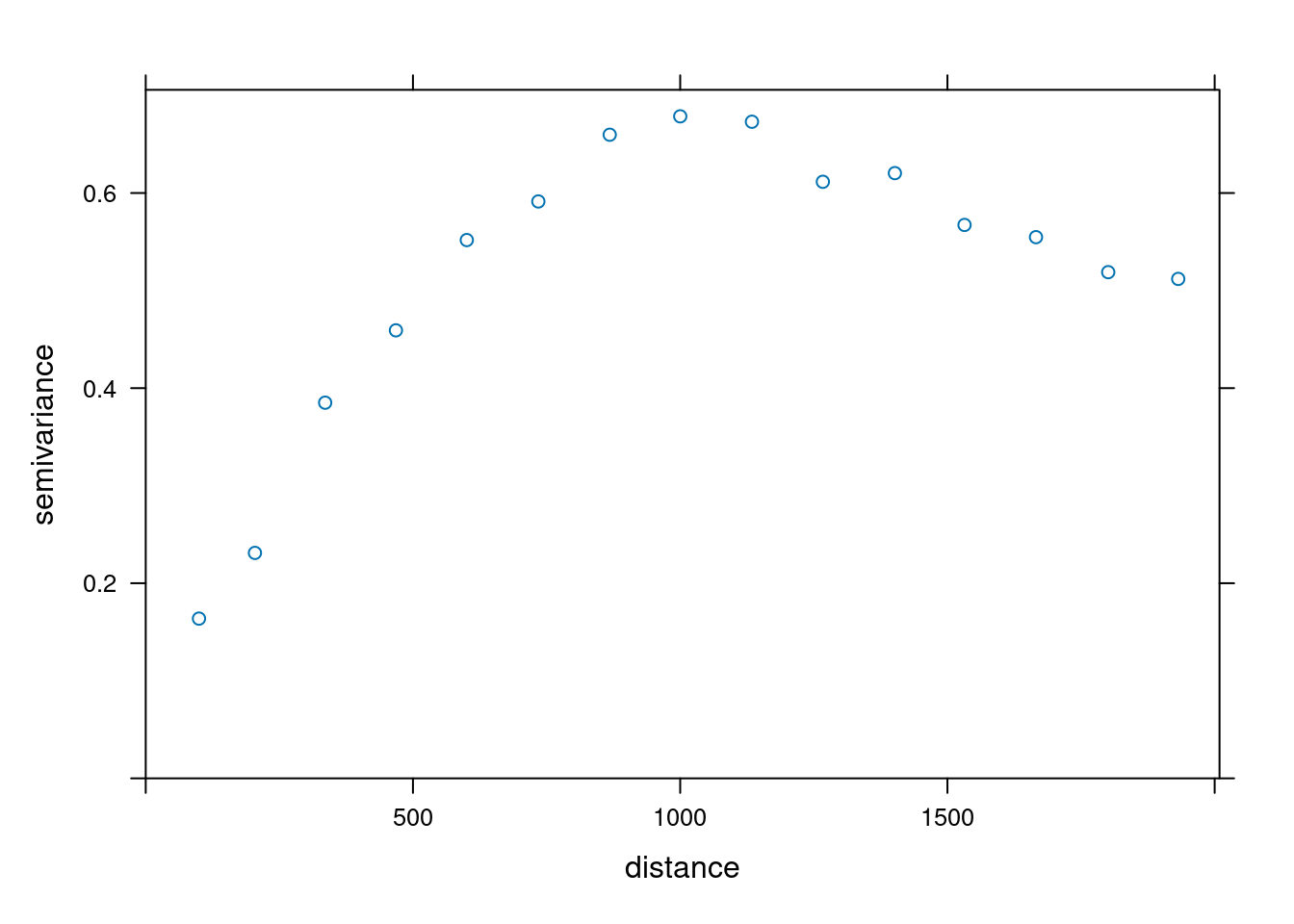
Le type vgm
Plusieurs modèles de variogrammes spatiaux sont disponibles dans la librairie.
show.vgms()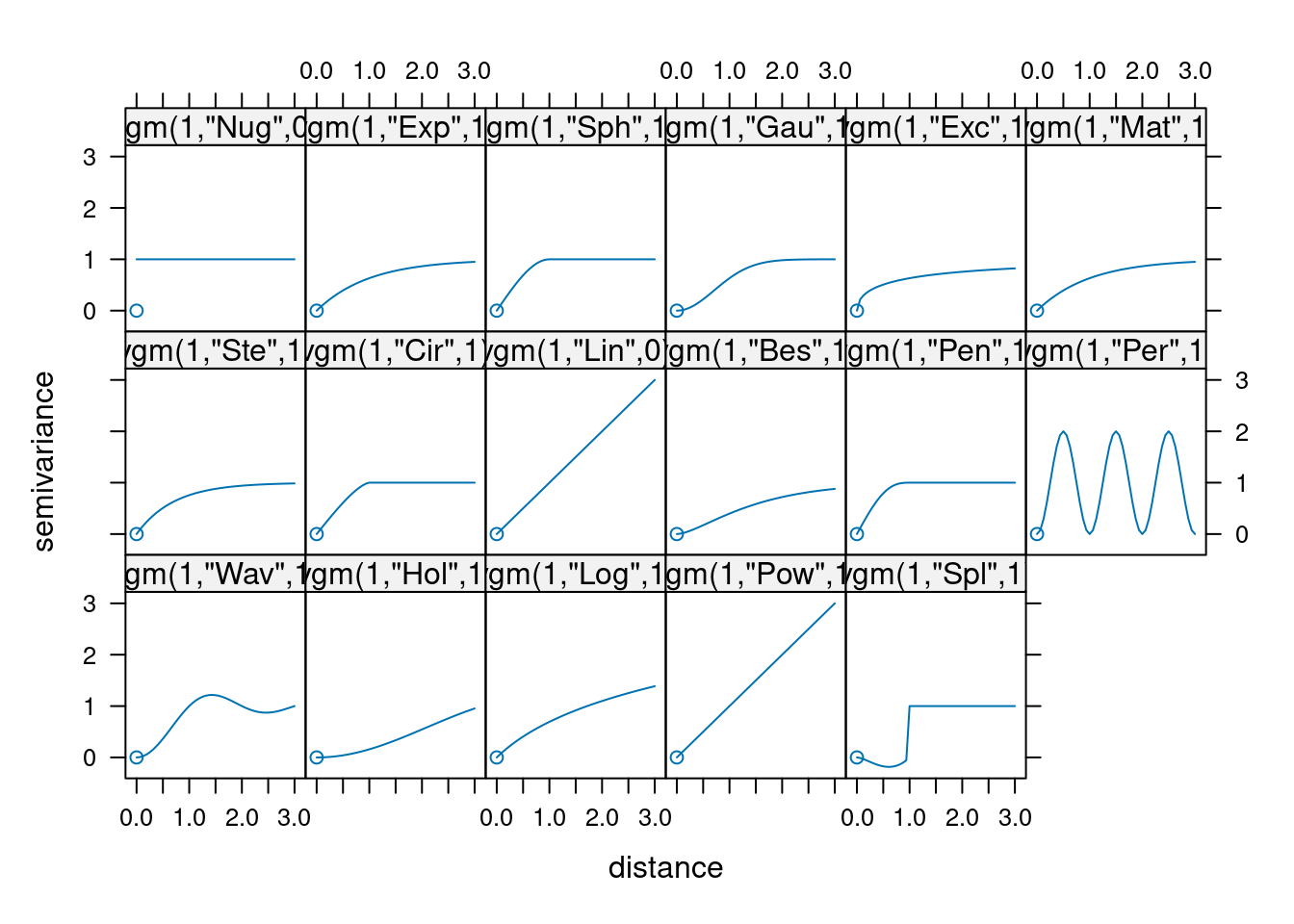
(vgm()) short long
1 Nug Nug (nugget)
2 Exp Exp (exponential)
3 Sph Sph (spherical)
4 Gau Gau (gaussian)
5 Exc Exclass (Exponential class/stable)
6 Mat Mat (Matern)
7 Ste Mat (Matern, M. Stein's parameterization)
8 Cir Cir (circular)
9 Lin Lin (linear)
10 Bes Bes (bessel)
11 Pen Pen (pentaspherical)
12 Per Per (periodic)
13 Wav Wav (wave)
14 Hol Hol (hole)
15 Log Log (logarithmic)
16 Pow Pow (power)
17 Spl Spl (spline)
18 Leg Leg (Legendre)
19 Err Err (Measurement error)
20 Int Int (Intercept)Un variogramme théorique est un objet de type vgm, qui doit être initialisé avec des paramètres avant de l’insérer dans la fonction d’estimation.
Un inconvénient dans la création d’un objet vgm est l’absence de nom pour certains paramètres, il faut se référer à la documentation pour savoir à quel place il faut rentrer quel argument lorsque’on appelle vgm(). Ne pas renseigner un argument initialise celui-ci à NA. Utiliser fit.variogram sur un objet avec des NA fait appel à une étape supplémentaire qui initialise avec des paramètres issus du variogramme empirique. Cela empêche de contrôler les valeurs initiales de l’estimation.
Estimation du variogramme théorique
Les paramètres du variogramme théorique sont ajustés par moindres carrés sur le variogramme empirique.
vgmModelwav <- fit.variogram(test,vgm(‘Wav’))
vgmModelexp <- fit.variogram(test, vgm(2, "Exp", 1000))
vgmModelexp model psill range
1 Exp 0.6849652 421.2255vgmModelmat <- fit.variogram(test, vgm(10, "Mat", 300, 4.5, kappa = 0.7))
vgmModelmat model psill range kappa
1 Nug 0.05112814 0.0000 0.0
2 Mat 0.62395526 340.0403 0.7vgmModelNA <- fit.variogram(test,vgm('Mat'))
vgmModelNA model psill range kappa
1 Nug 0.001864289 0.0000 0.0
2 Mat 0.684275121 424.4234 0.5start_time <- Sys.time()
vgmModelwav <- fit.variogram(test,vgm('Wav'))
end_time <- Sys.time()
end_time - start_timeTime difference of 0.004230022 secsvgmModelwav model psill range
1 Nug 0.1556503 0.0000
2 Wav 0.4407740 643.9395La fonction plot() appelée sur un modèle estimé trace en continu le variogramme théorique et en points le variogramme empirique.
plot(test, vgmModelmat)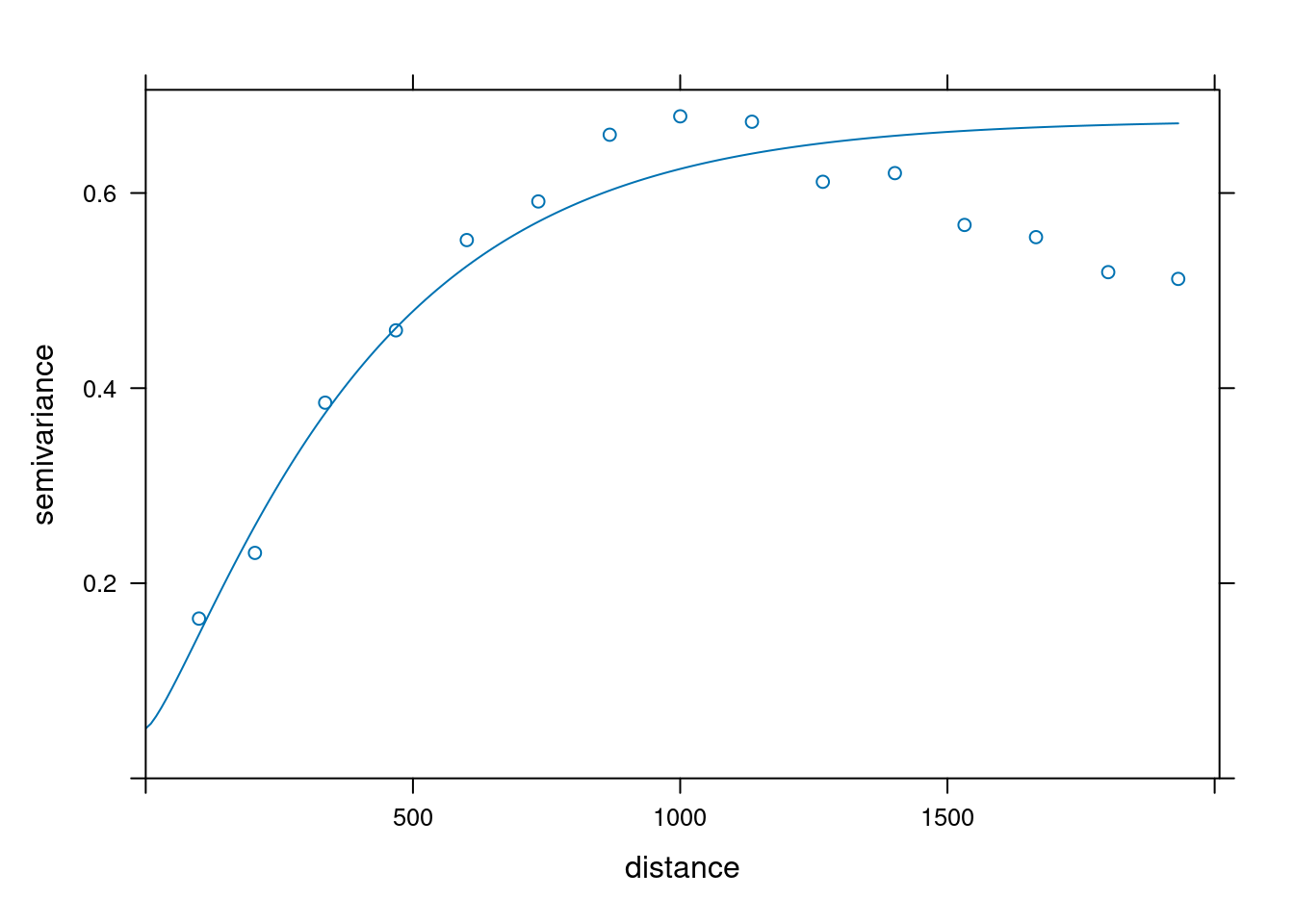
plot(test, vgmModelwav)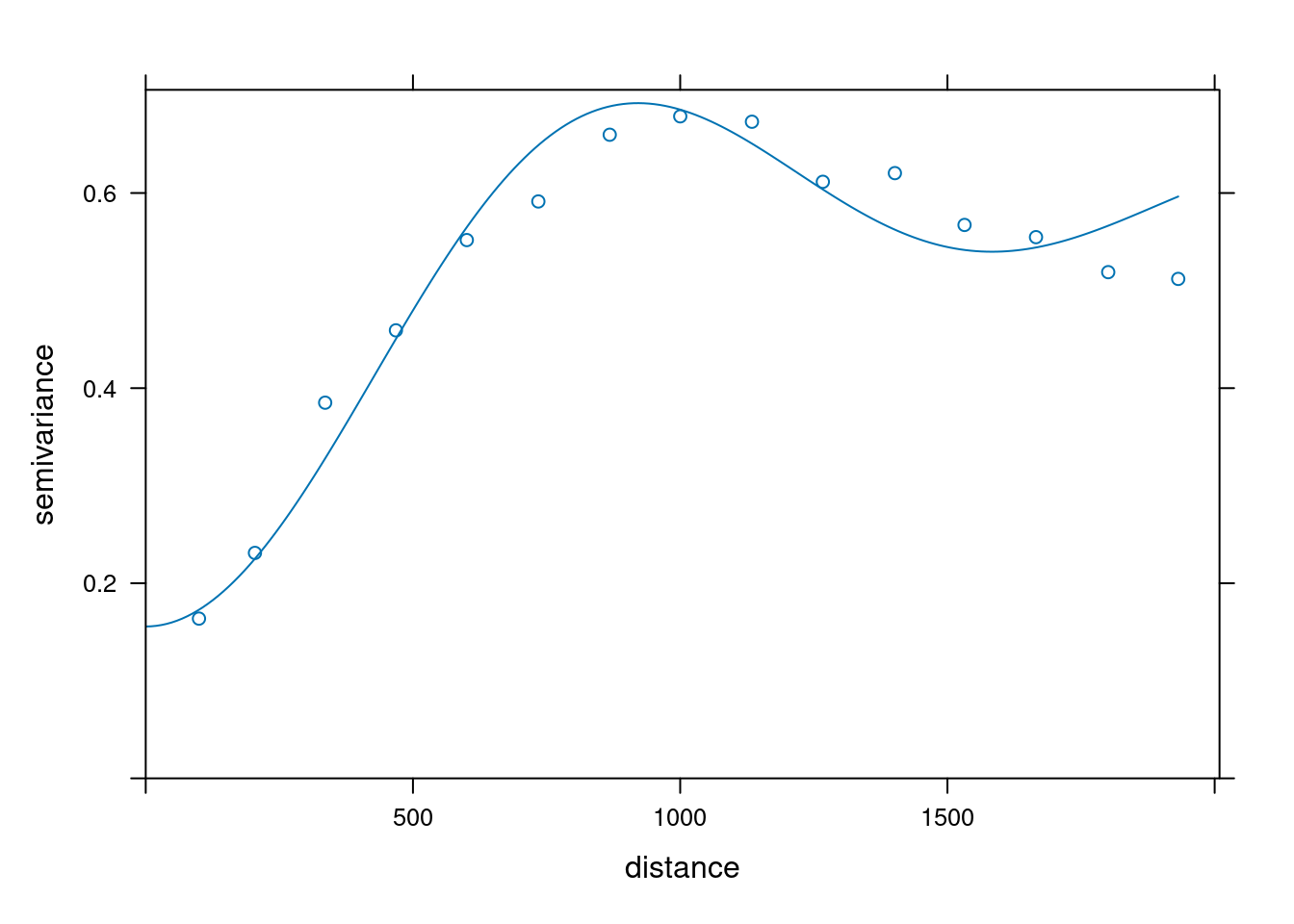
Krigeage
start_time <- Sys.time()
krig <- krige(log(zinc)~1 , df_sf, df_grid_sf, model = vgmModelmat)[using ordinary kriging]end_time <- Sys.time()
end_time - start_timeTime difference of 0.5332577 secsKres_pred = data.frame(x=meuse.grid$x,y=meuse.grid$y,pred=krig$var1.pred)
krig_res<- terra::rast(Kres_pred, type="xyz", crs="EPSG:2154")
Kres_var = data.frame(x=meuse.grid$x,y=meuse.grid$y,pred=krig$var1.var)
krig_var<- terra::rast(Kres_var, type="xyz", crs="EPSG:2154")
plot((krig_res), main = 'Krigeage de log(zinc) - gstat')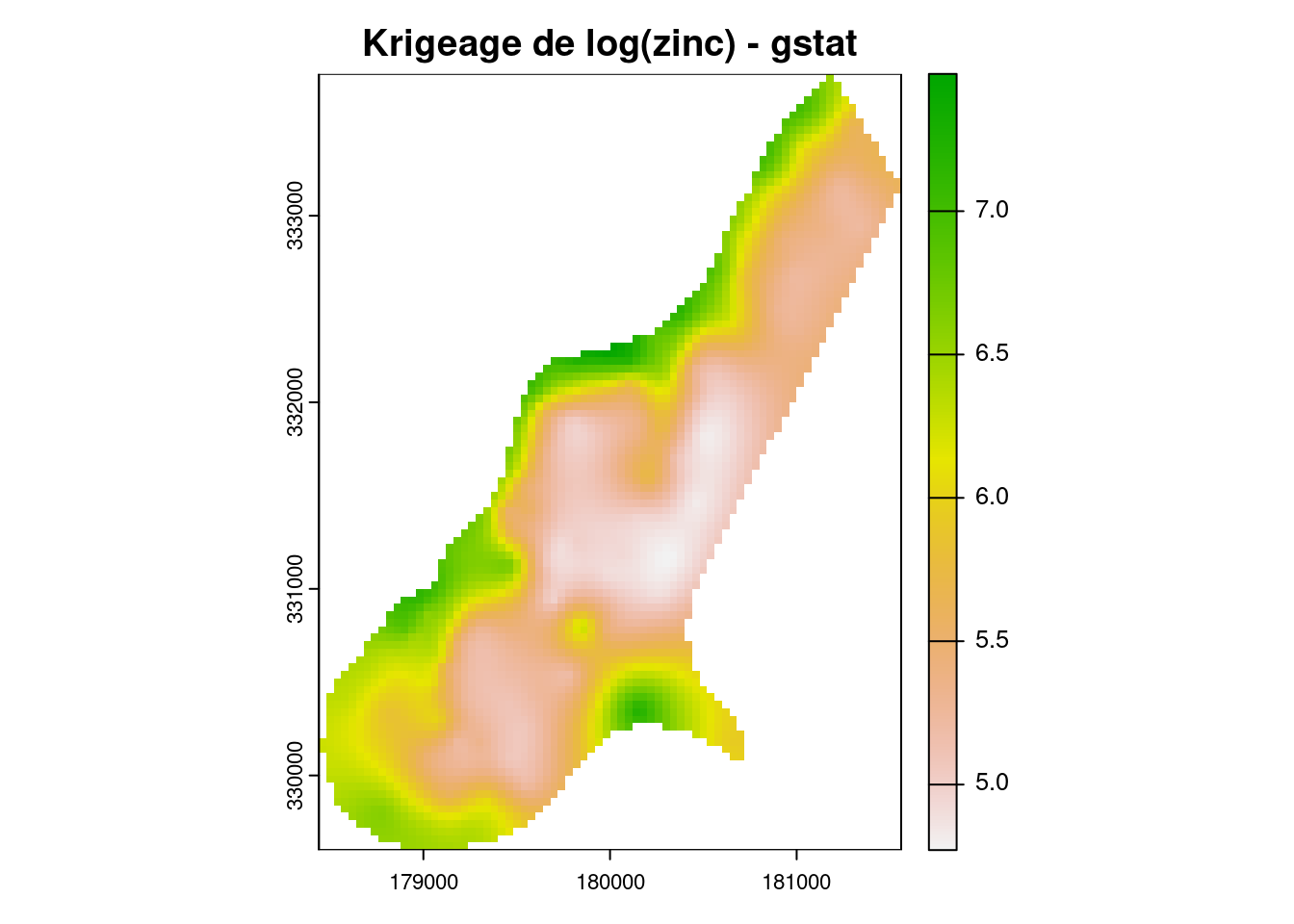
plot(krig_var,main = 'Erreur de krigeage pour log(zinc) - gstat')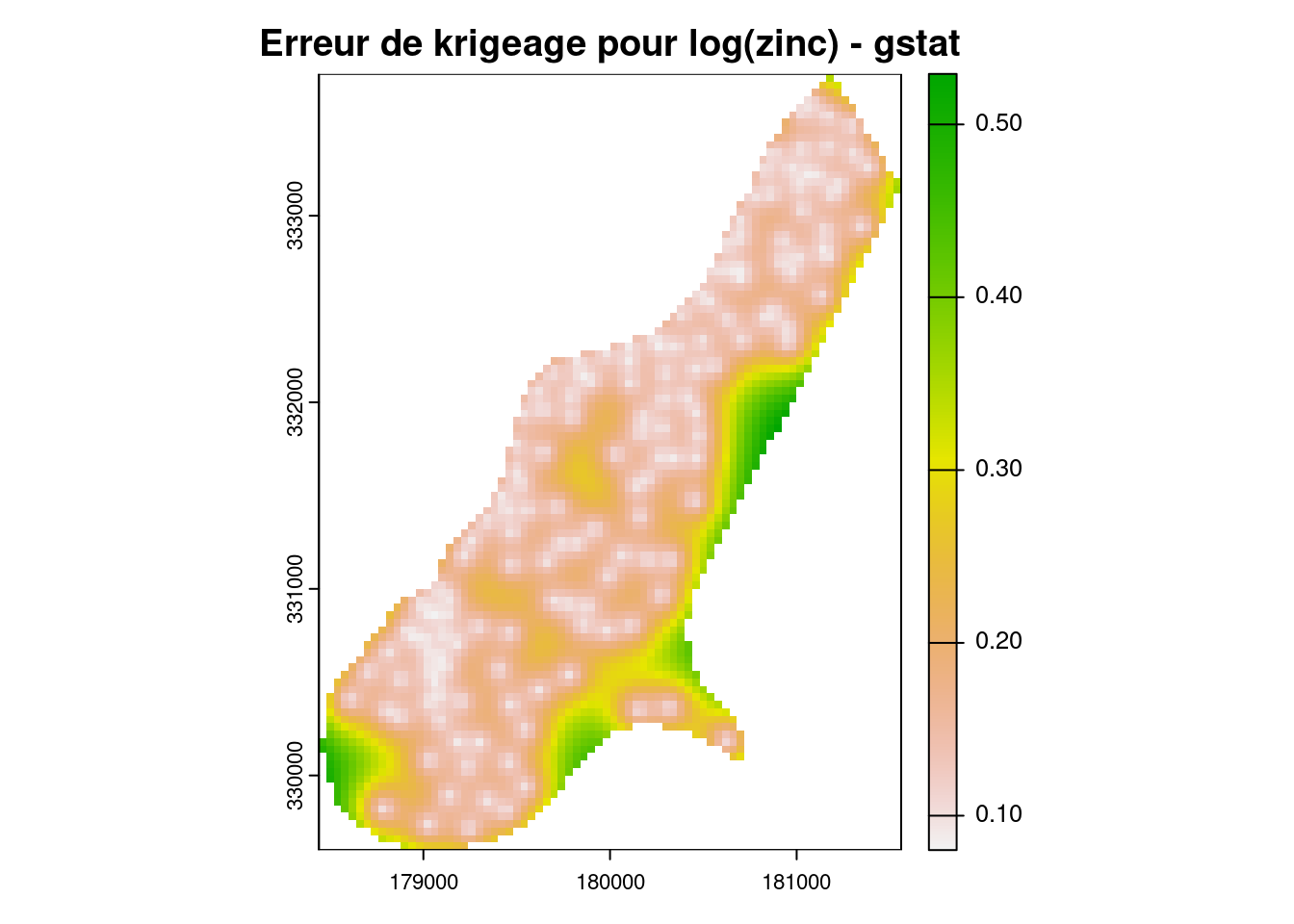
2 - Package R GeoModels
GeoModels est un package relativement récent (2022), il prend la suite de CompRandFld suite à une mise à jour majeure de R.
Il est disponible sur le CRAN :
https://CRAN.R-project.org/package=GeoModels
Le package propose des fonctions pour de la simulation, de l’inférence et de la prédiction de champs spatiaux et spatio-temporels, univarié ou multivarié. Il contient des méthode spour des champs Gaussiens et non Gaussiens.
On peut travailler en coordonnées euclidiennes et sphériques.
GeoModels propose de faire des modèles spatiaux et spatiotemporels.
Les estimations se font par maximisation de vraisemblance composite.
Du fait de la variété de modèles et de données possibles en entrée (spatial, spatiotemporel, 1 ou plusieurs variables …), chaque fonction est définie généralement pour prendre en entrée des données (notamment data et coordonnées) sous plusieurs types. Dans tous les cas, on ne dépend pas de structures “évoluées”, on n’a besoin que de vecteurs et de matrices. En contrepartie, on est probablement moins libres sur les systèmes de coordonnées.
Les données en entrée doivent être, si on fait du spatial uniquement :
- data : un vecteur de taille \(d\)
- coordx : une matrice \(d \times 2\) si on met directement les coordonnées, ou un vecteur de taille \(d\) si on sépare les coordonnées
- coordy : NULL si on a déjà mis les 2 coordonnées dans coordx, vecteur de taille \(d\) sinon.
Il n’y a donc pas besoin de gérer des types de données spatiales comme dans gstat.
data =log(df$zinc)
coordx = df$x
coordy = df$yVariogramme empirique
# Variogramme empirique :
start_time <- Sys.time()
emp_vario = GeoVariogram(data, coordx, coordy=coordy,numbins = 16,maxdist = 2000)
end_time <- Sys.time()
end_time - start_timeTime difference of 0.009788275 secsplot(emp_vario,main = "Variogramme empirique Zinc - GeoModels")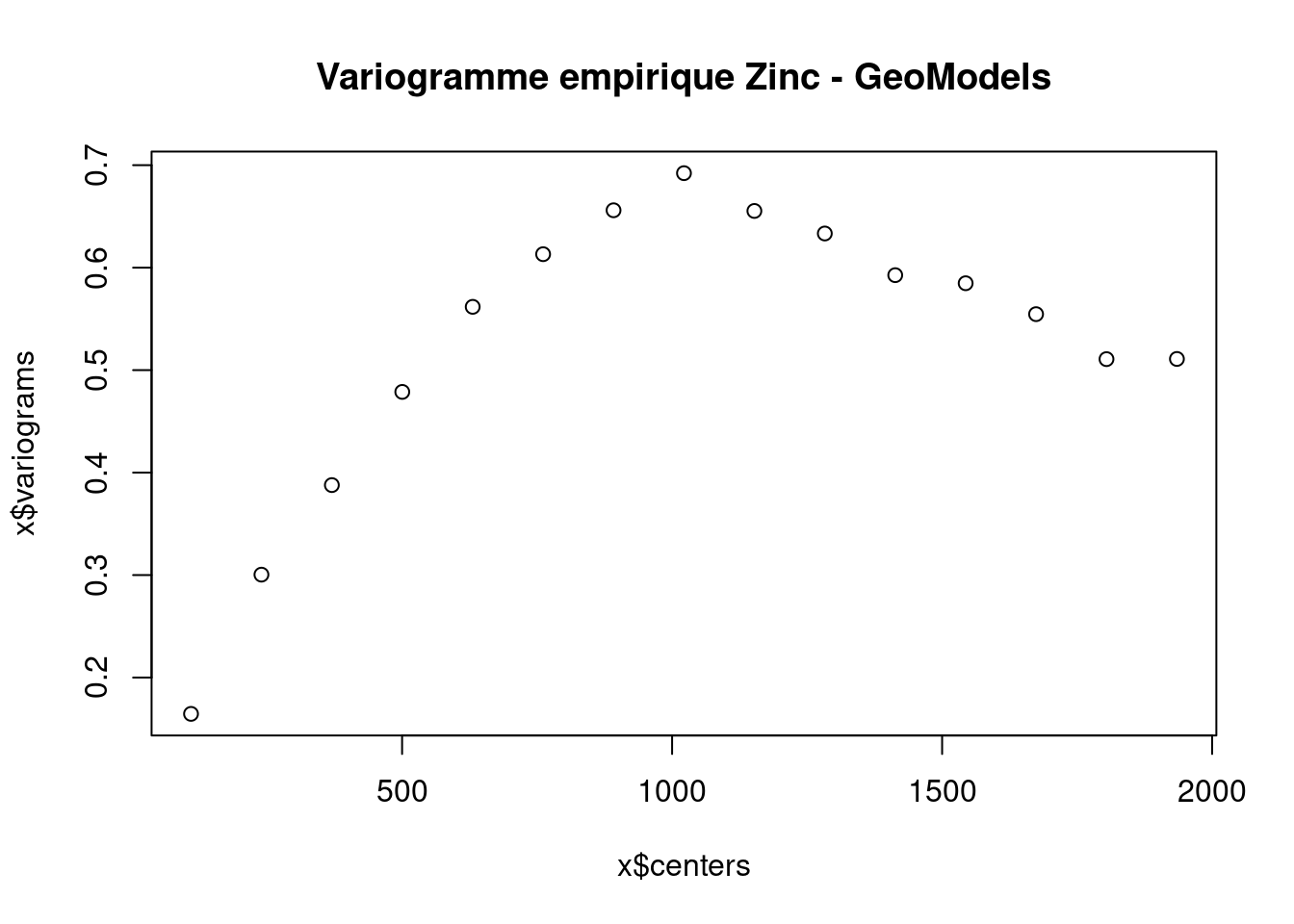
Modèles supportés
Voir documentation, fonction “GeoCovMatrix”
CorrParam, et NuisParam permettent d’obtenir la listes des paramètres requis pour les modèles.
NuisParam('Gaussian')[1] "mean" "nugget" "sill" CorrParam("Exp")[1] "scale"CorrParam('Matern')[1] "scale" "smooth"Estimation du variogramme théorique
L’estimation nécessite de donner en entrée deux listes, fixed et start, correspondant aux paramètres fixés et libres dans l’ajustement.
ATTENTION : si on ne fixe rien, il ne faut pas une liste vide mais un NULL, ou alors ne pas renseigner l’argument (NULL est la valeur par défaut)
L’algorithme vérifiera automatiquement que tous les paramètres nécessaires sont remplis. Il n’y a pas comme dans gstat de méthode qui rempli automatiquement des oublis, il faut vérifier avec les fonctions précédentes qu’on a bien tout initialisé.
fixed = NULL
start = list( mean = 0 , sill=0.5,smooth=0.7,scale = 300,nugget = 0.044)
modelemat = GeoFit(data, coordx=coordx, coordy=coordy, fixed = fixed, start=start,
corrmodel="Matern",distance="Eucl",maxdist = 2000)
modelemat
##################################################################
Maximum Composite-Likelihood Fitting of Gaussian Random Fields
Setting: Marginal Composite-Likelihood
Model: Gaussian
Type of the likelihood objects: Pairwise
Covariance model: Matern
Optimizer: Nelder-Mead
Number of spatial coordinates: 155
Number of dependent temporal realisations: 1
Type of the random field: univariate
Number of estimated parameters: 5
Type of convergence: Successful
Maximum log-Composite-Likelihood value: -37443.52
Estimated parameters:
mean nugget scale sill smooth
5.8692 0.9994 266.5697 0.5482 68.5193
################################################################### Vario fit : il faut mettre en entrée des valeurs de départ et des valeurs qu'on fixe pour les paramètres
fixed = list(nugget = 0.044) #fixe la valeur obtenue avec gstat
start = list( mean = 0 , sill=0.5,smooth=0.7,scale = 300)
modelemat2 = GeoFit(data, coordx=coordx, coordy=coordy, fixed = fixed, start=start,
corrmodel="Matern",distance="Eucl",maxdist = 2000)
modelemat2
##################################################################
Maximum Composite-Likelihood Fitting of Gaussian Random Fields
Setting: Marginal Composite-Likelihood
Model: Gaussian
Type of the likelihood objects: Pairwise
Covariance model: Matern
Optimizer: Nelder-Mead
Number of spatial coordinates: 155
Number of dependent temporal realisations: 1
Type of the random field: univariate
Number of estimated parameters: 4
Type of convergence: Successful
Maximum log-Composite-Likelihood value: -37218.17
Estimated parameters:
mean scale sill smooth
5.8673 114.4160 0.5495 1.5971
##################################################################fixed = NULL
start = list( mean = 0 , sill=0.5,scale = 300,nugget = 0.044)
start_time <- Sys.time()
modelewav = GeoFit(data, coordx=coordx, coordy=coordy, fixed = fixed, start=start,
corrmodel="Wave",distance="Eucl",maxdist = 2000)
end_time <- Sys.time()
end_time - start_timeTime difference of 0.755408 secsmodelewav
##################################################################
Maximum Composite-Likelihood Fitting of Gaussian Random Fields
Setting: Marginal Composite-Likelihood
Model: Gaussian
Type of the likelihood objects: Pairwise
Covariance model: Wave
Optimizer: Nelder-Mead
Number of spatial coordinates: 155
Number of dependent temporal realisations: 1
Type of the random field: univariate
Number of estimated parameters: 4
Type of convergence: Successful
Maximum log-Composite-Likelihood value: -37191.31
Estimated parameters:
mean nugget scale sill
5.8669 0.3839 200.4165 0.5509
##################################################################On peut comparer les modèles que l’on vient de faire avec le variogramme empirique :
# Comparaison avec l'empirique matern :
GeoCovariogram(modelemat, distance="Eucl",show.vario= TRUE,vario = emp_vario)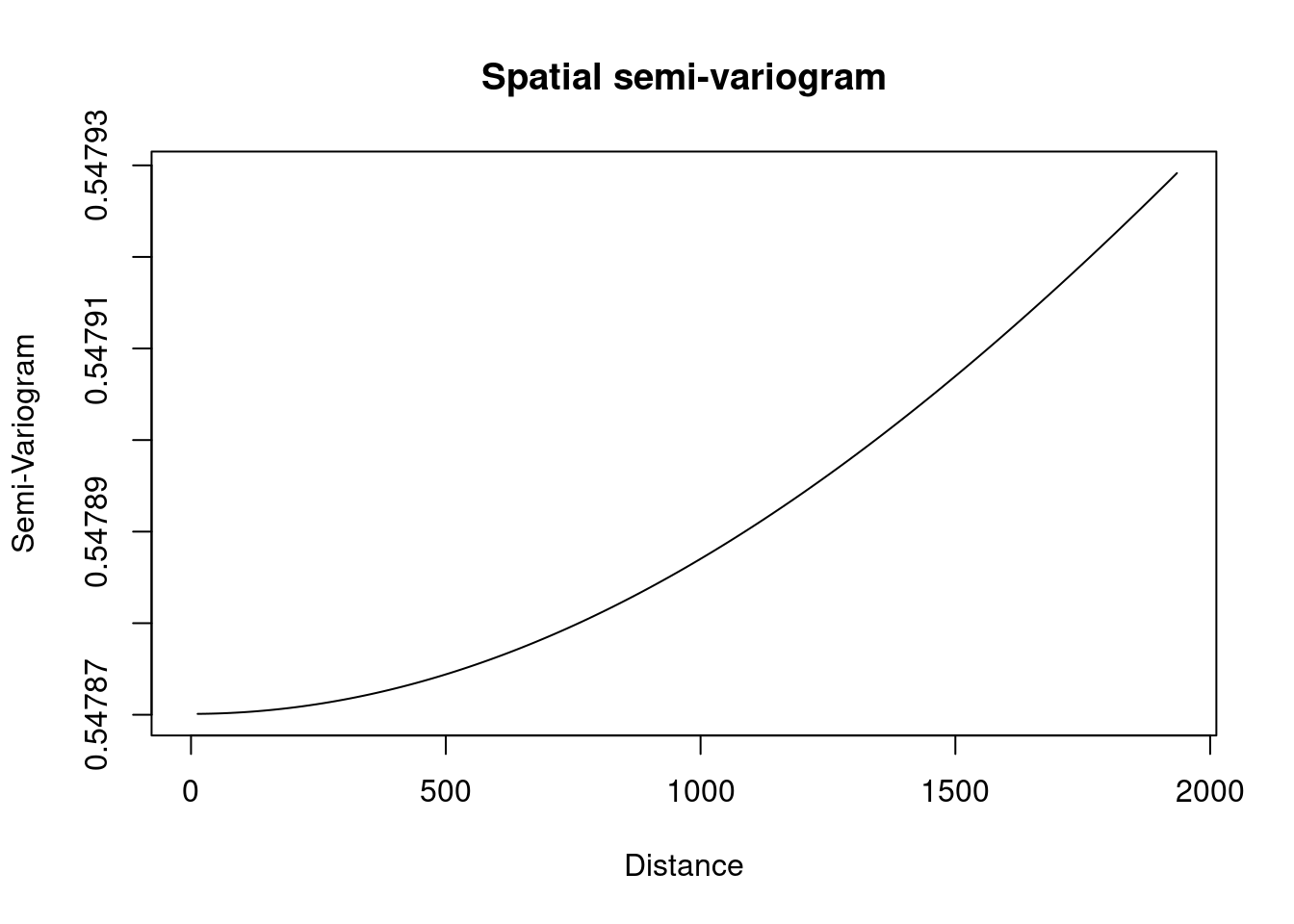
GeoCovariogram(modelemat2, distance="Eucl",show.vario= TRUE,vario = emp_vario)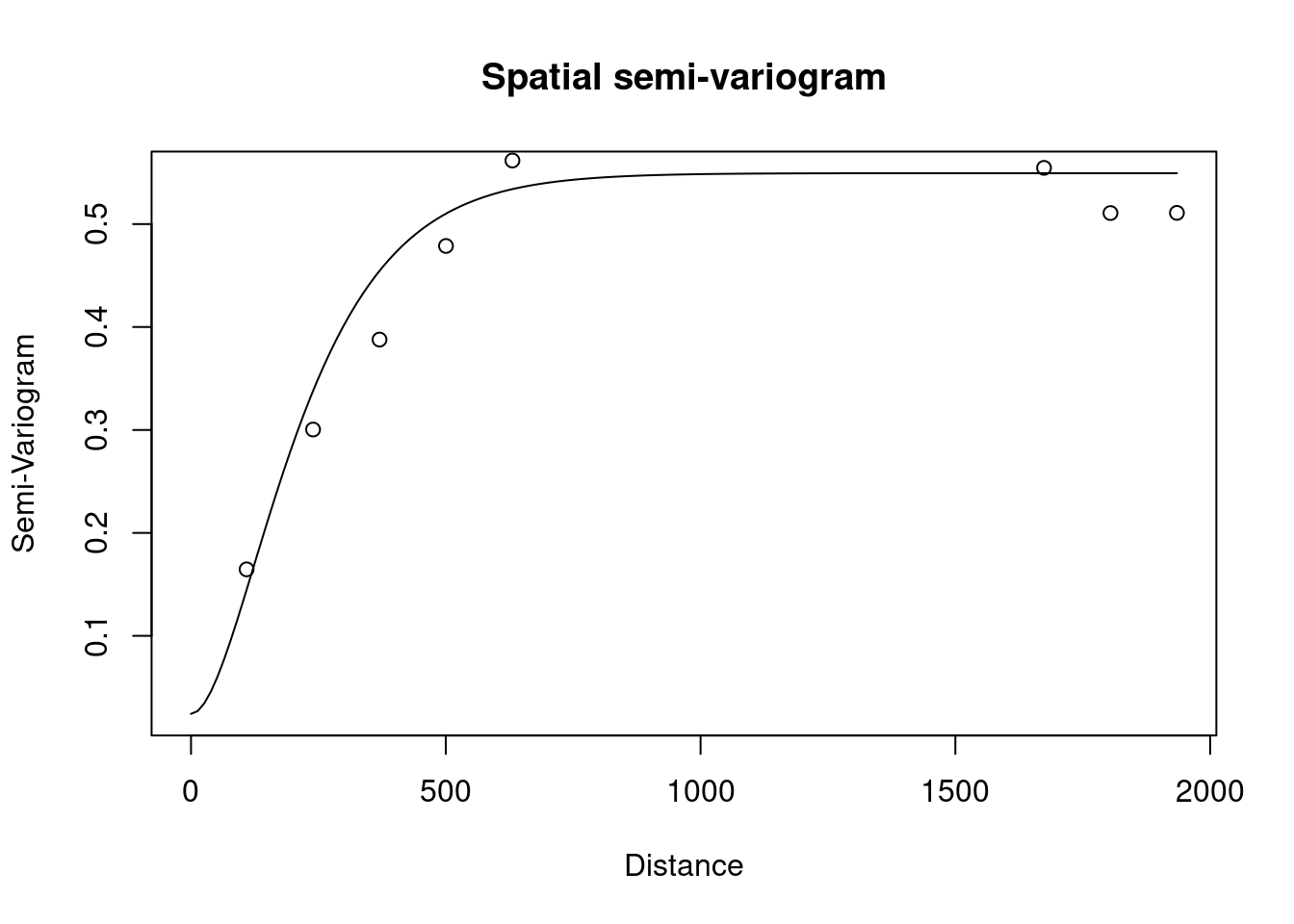
# Comparaison avec l'empirique Wave :
GeoCovariogram(modelewav, distance="Eucl",show.vario= TRUE,vario = emp_vario)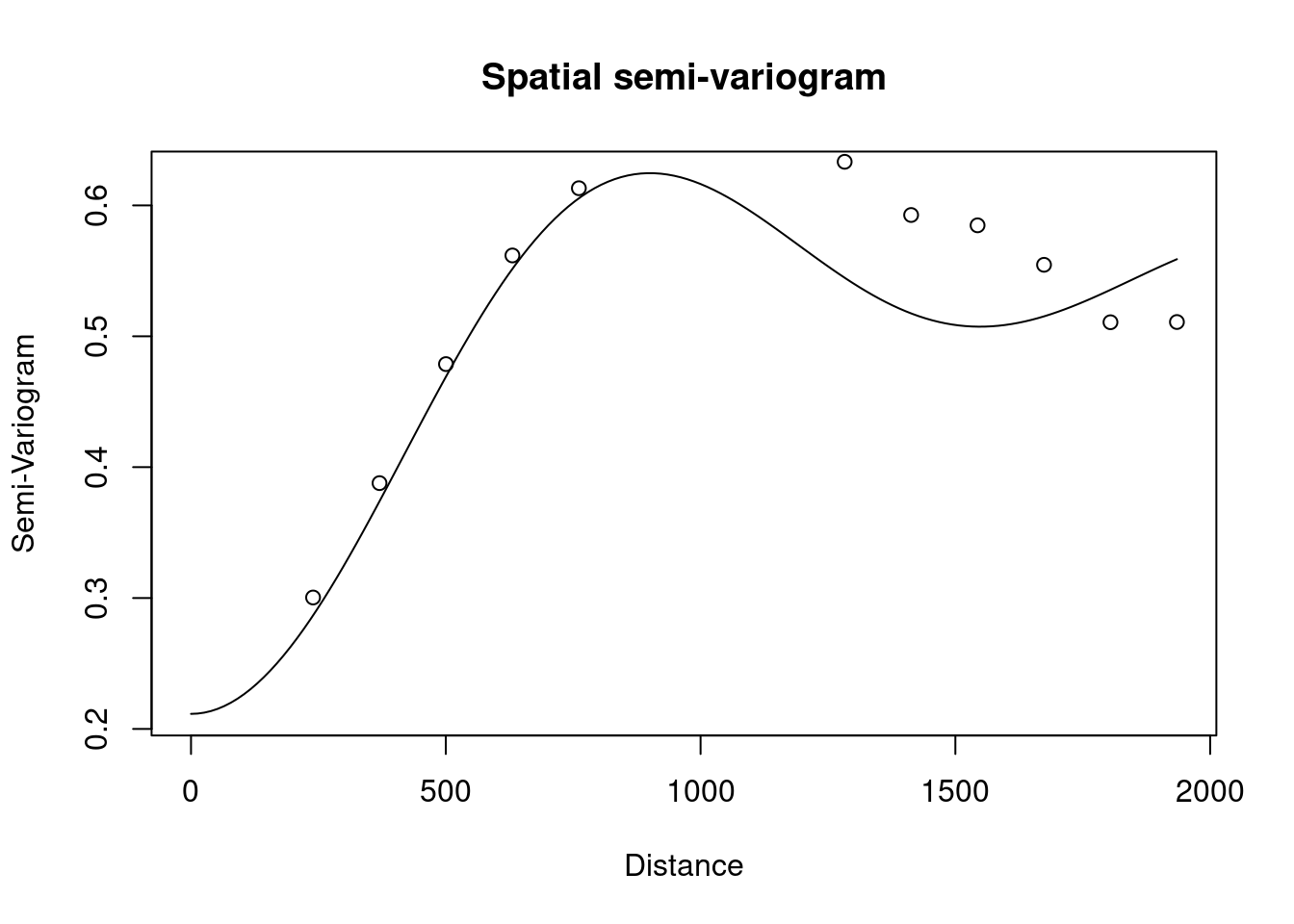
Krigeage
locskrig = data.frame (x =meuse.grid$x,y= meuse.grid$y)
start_time <- Sys.time()
krig_geomodels = GeoKrig(loc= locskrig,coordx=coordx, coordy=coordy,corrmodel = 'Wave',param = c(modelemat$fixed,modelewav$param),mse =TRUE,data=data)
end_time <- Sys.time()
end_time - start_timeTime difference of 0.7958126 secscoords = cbind(coordx,coordy)
Kres_pred_geo = data.frame(x=meuse.grid$x,y=meuse.grid$y,pred=krig_geomodels$pred)
krig_res_geo<- terra::rast(Kres_pred_geo, type="xyz", crs="EPSG:2154")
Kres_var_geo = data.frame(x=meuse.grid$x,y=meuse.grid$y,pred=krig_geomodels$mse)
krig_var_geo<- terra::rast(Kres_var_geo, type="xyz", crs="EPSG:2154")
plot((krig_res_geo), main = 'Krigeage de log(zinc) - GeoModels')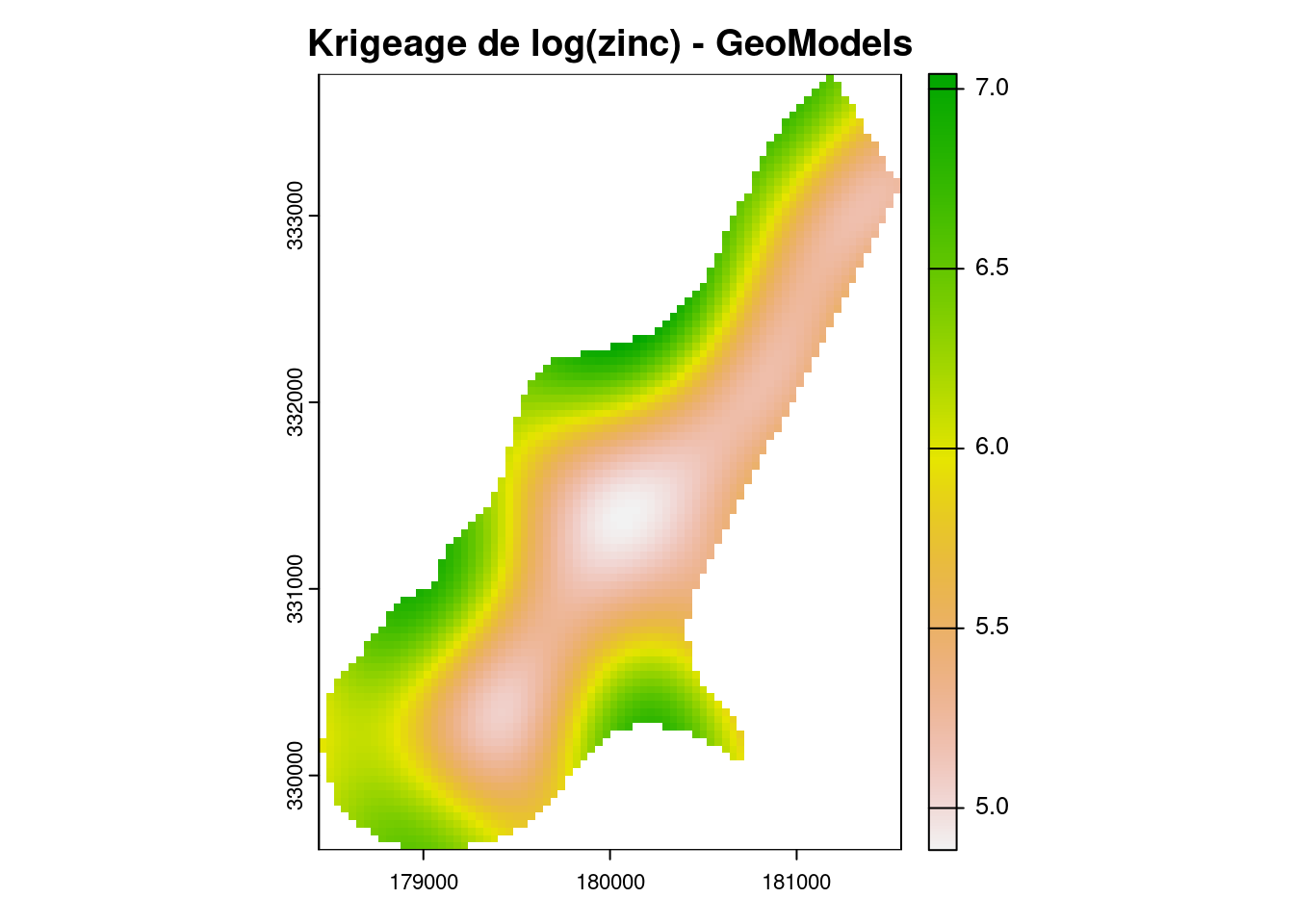
plot(krig_var_geo,main = 'Erreur de krigeage pour log(zinc) - GeoModels')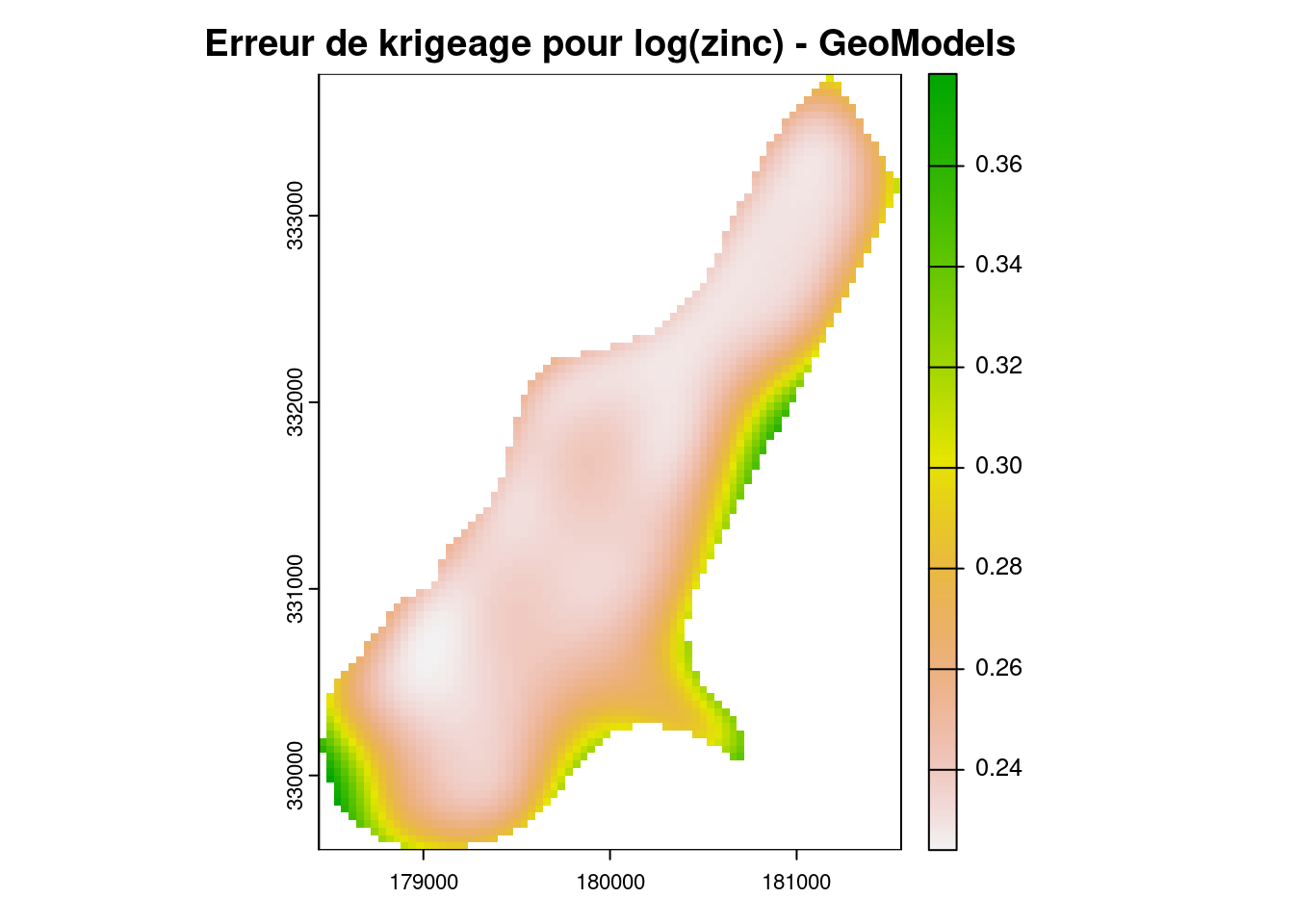
3 - Package Julia GeoStats
Julia est un langage de programmation de haut niveau, performant et dynamique pour le calcul scientifique, avec une syntaxe familière aux utilisateurs d’autres environnements de développement similaires (Matlab, R, Scilab, Python, etc.). Il fournit un compilateur sophistiqué, un système de types dynamiques avec polymorphisme paramétré, une exécution parallèle distribuée, des appels directs de fonctions C, Fortran et Python. (source Wikipedia)
Un avantage de Julia est la possibilité d’écrire en LaTeX dans les lignes de code.
GeoStats est un package récent (avril 2015), sa dernière mise à jour date de 3 mois, c’est le package majeur en statistiques spatiales de Julia.
Il est disponible sur le :
https://juliaearth.github.io/GeoStats.jl/stable/
Pour l’installer faire la commande suivante dans le terminal de votre IDE : ]add GeoStats
Le package propose des fonctions pour de la simulation, de l’inférence et de la prédiction de champs spatiaux
On peut travailler en coordonnées euclidiennes et sphériques.
GeoStats propose de faire des modèles spatiaux. Sa particularité va être de tester tous les variogrammes disponibles (dans une même exécution) dans le package pour obtenir le plus optimisé avec les paramètres qui fonctionnent le mieux.
Bibliothèques Julia
# Import package
using GeoDataFrames, Distributed, StatsAPI, Plots, DelimitedFiles, DataFrames, CSV, GeoStats, Rasters, Shapefile,GeoTables, CairoMakie; const GDF=GeoDataFrames; import WGLMakie as MkeImporter les données
# Get the current working directory
current_directory = pwd()
#import data
meuse = CSV.read(joinpath(current_directory,"meuse.txt"),DataFrame;delim="," )
meuse_grid = CSV.read(joinpath(current_directory, "meuse.grid.txt"),DataFrame; delim=",")
meuse_geom = georef(CSV.File(joinpath(current_directory,"meuse.txt")),(:x,:y,:cadmium))
#describe data
describe(meuse)
describe(meuse_grid)Transformation en données spatiale
#add log zinc to the data
logmeuse = log.(meuse.zinc)
meuse.logzinc = logmeuse
#create coord of data meuse
coord = [(x, y) for (x, y) in zip(meuse.x, meuse.y)]
#create spatial object of meuse
meuselog = georef(meuse[:,["x","y","logzinc"]],coord)
# list of properties with coordinates
props = (logzinc=meuse.logzinc,)
𝒟 = georef(props, coord)Variogramme
Le fit pour tester les différents variogramme va nous donner le SineHoleVariogram avec tous les paramètres optimisé. On décide à la base d’utiliser ce dernier mais il s’avère qu’il ne donne pas de bon résultat pour le Krigeage. On utilisera donc un variogramme de Matérn. Si vous voulez observer ce dernier dans le krigeage il suffira de mettre dans la fonction kriging, le variogramme désiré.
# empirical variogram
@time empvario = EmpiricalVariogram(meuselog, :logzinc, maxlag = 2000.)
## plot of empirical variogram
# Mke.plot(empvario) plot interactive
# CairoMakie.plot(empvario) #plot sans interaction
## plot theorical variogram
# CairoMakie.plot(MaternVariogram(range=691, nugget=0.04))
# CairoMakie.plot(SineHoleVariogram(range=691, nugget=0.25))
#fit theorical and empirical variogramand
@time fitvario = fit(Variogram, empvario)
# CairoMakie.plot(empvario)
# CairoMakie.plot!(fitvario, maxlag = 2000.)
# CairoMakie.current_figure()Création de la grille
# grid creation
# Convert the x and y coordinates to numeric arrays
x_coords = Float64.(meuse.x)
y_coords = Float64.(meuse.y)# Create the Cartesian grid dimensions, origin, and spacing
dims = (100, 100) # Number of grid points in x and y directions
origin = (minimum(x_coords), minimum(y_coords)) # Lower left corner of the grid
spacing = ((maximum(x_coords) - minimum(x_coords)) / (dims[1] - 1),
(maximum(y_coords) - minimum(y_coords)) / (dims[2] - 1))
# Cell spacing in x and y directions# Create the CartesianGrid
𝒢 = CartesianGrid(dims, origin, spacing)Krigeage
𝒫 = EstimationProblem(𝒟, 𝒢, :logzinc)
@time 𝒮 = Kriging(:logzinc => (variogram=MaternVariogram(nugget = 0.21,sill = 0.59,range=706.78),))
# perform estimation
@time Ω = solve(𝒫, 𝒮)
# CairoMakie.plot(Ω.geometry, color = Ω.logzinc_variance)Affichage du Krigeage
#Export Kriging
GeoTables.save(joinpath(current_directory,"data_geostats_julia/Kriging2.shp"), Ω,force=true )
#read kriging
shape = Shapefile.Handle(joinpath(current_directory,"data_geostats_julia/Kriging2.shp")).shapes
# read grid
grid_shp = Shapefile.Handle(joinpath(current_directory,"data_geostats_julia/boundary1.shp")).shapes
# Define a reducer function (e.g., to sum values within each cell)
reducer(cell_values) = sum(cell_values)
rasterized_krig = rasterize(shape, fill=Ω.logzinc, reducer = reducer, size=(100, 100))
Plots.plot(rasterized_krig)Burning each geometry to a BitArray slice... 0%| | ETA: 0:48:28
Burning each geometry to a BitArray slice... 100%|██████████████████████████████████████████████████| Time: 0:00:00
Reducing... 4%|██ | ETA: 0:00:02
Reducing... 47%|███████████████████████▌ | ETA: 0:00:00
Reducing... 89%|████████████████████████████████████████████▌ | ETA: 0:00:00
Reducing... 100%|██████████████████████████████████████████████████| Time: 0:00:00
#Crop
masked = mask(rasterized_krig,with=grid_shp)
Plots.plot(masked, title = "Krigeage logzinc")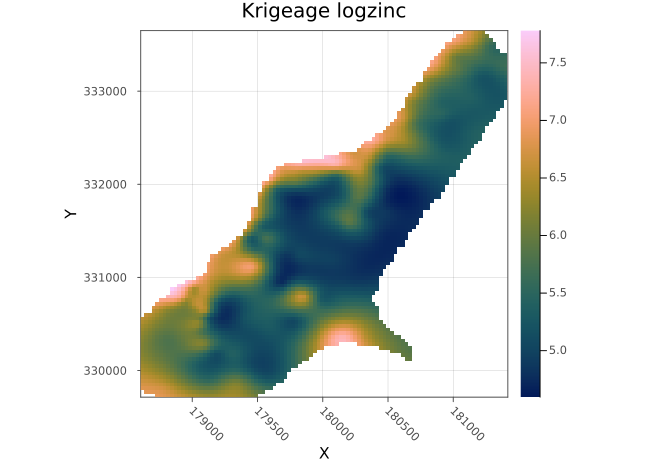
4 - Comparaison
| Package | R : gstat | R : GeoModels | Julia : GeoStats |
|---|---|---|---|
| Type de données d’entrée | sp, sf | matrices, vecteurs |
dataframes Coordonées de type CartesianGrid Inconvénient : peu de documentation - difficile à prendre en main |
| Documentation | CRAN |
CRAN https://vmoprojs.github.io/GeoModels-page/ |
https://github.com/JuliaEarth/GeoStats.jl https://juliapackages.com/p/geostats |
| Estimation de paramètres du variogramme |
Moindres carrés sur variogramme empirique 1 modèle à la fois au choix |
Vraisemblance composite sur les données 1 modèle à la fois Paramètres fixes ou variables au choix |
Moindres carrés sur le variogramme empirique Tous les modèles disponibles à la fois |
| Vitesse d’exécution - estimation du variogramme empirique | 0.05s | 0.07s | 0.012s |
| Vitesse d’exécution - estimation du variogramme théorique | 0.04s | 1.15s | 0.05s (TOUS LES MODELES) |
| Vitesse d’exécution - krigeage | 0.42s | 0.87s |
0.08s (Kriging) 10.6s (solve) |