Personalisation de Rmarkdown
Base de fonctionnement de Rmarkdown
Résumé de la chaîne de traitement
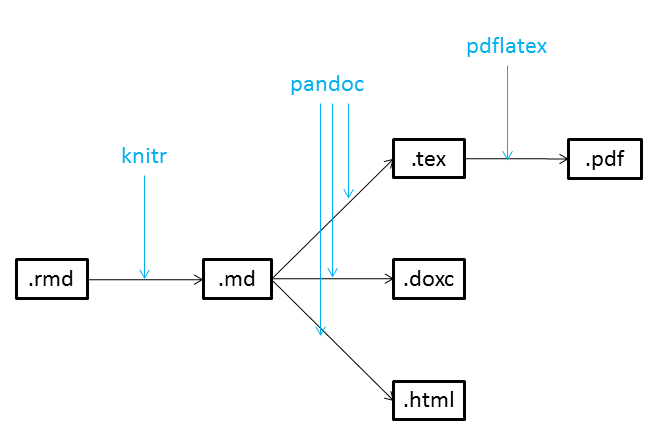
Process de création d’un document
Les acteurs
- Chaîne de traitement
- package/fonction rmarkdown: gestion de l’ensemble de la chaîne de traitement (Rstudio -> document final)
- package knitr: interprétation du code utilisateur (R/Python/Julia) et conversion en sortie intégrable au document
- logiciel Pandoc: convertisseur de markdown (faiblement structuré) en langage balisé (fortement structuré)
- Interpréteur de langage balisé
- navigateur web (HTML)
- commande
(pdf)latex+ lecteur (PDF) - Word/libreoffice (docx)
Langages
- Metadata (YAML): paramétrisation de la chaîne de traitement
- Langage pour code utilisateur (R, Python, Julia, bash…)
- Langage de balisage (Rmarkdown, markdown, HTML, \(\LaTeX2e\), XML)
Organisation d’un document structuré par balises
- Un préambule
- preamble (précède
\begin{document}) dans \(\LaTeX\) - une en-tête en
HTML(<header> ... <\header>)
- preamble (précède
- document principal
\begin{document} ... \end{document}en \(\LaTeX\)<body> ... </body>enHTML
Fichiers
- entrée
- .Rmd (YAML+R+markdown)
- fichier intermédiare
- .md (markdown), figures
- configuration
- .sty, .css
- sortie
- .tex/.PDF, .html, .docx
Fonctionnement du préambule Rmd
L’idée est de transformer le Rmd en md et qu’il soit interprété par pandoc. Les options vont donc devoir être passéees à knitr et à Pandoc. On a donc un préambule pour cela. Néanmoins on voit à ce moment de l’histoire que les options vont être traitées différemment selon les étapes. Il faut donc récupérer les options sous une forme structurée et non plus balisée. C’est le but de yaml qui est un parseur. Ceci explique aussi pourquoi l’indentation et la présentation sont importantes. Les éléments parsés vont être transmis aux interpréteurs.
Notez que le type de sortie (html_document, pdf_document) correspond à une fonction du package rmarkdown (html_document() ou pdf_document()) dont la tâche est d’effectuer toute la chaîne de traitement. Les entrées YAML associé par l’indentitation à cette fonction correspondent en faite à des variables passés en arguments de cette fonction paramétrant le processus.
On peut distinguer les options globales des options qui sont spécifiques d’un format.
Options globales
Elles sont communes à tout type de sortie
---
title: titre du document
subtitle: "sous titre"
author: auteur du document
date: "`r Sys.Date()`"
---Options spécifiques
On peut distinguer pour HTML ou les fichiers de définition inclus (css ou .sty) ou ou l’utilisation de certaines commandes (on donne une valeur à une variable).
Exemple pour html_document()
---
title: titre du document
author: auteur du document
output:
html_document:
number_sections: yes
toc: true
mathjax: "http://example.com/MathJax.js"
css: monstyle.css
---Exemple pour pdf_document()
---
title: titre du document
author: auteur du document
output:
pdf_document:
number_sections: yes
toc: true
include:
in_header: preamble.sty
---Example avancé pour la sortie beamer_presentation
Créer son propre template Rmd
Si la customisation du document est importante il devient vite illisible de les inclure en paramètres dans le préambule et il faut mieux créer son propre template. Ceci revient à créer une libraire R un peu spéciale et l’inclure.
Exemple: un package pour une publication scientifique
Un exemple simple est le template d’un journal. Il faut créer package qui s’organisera comme dans la figure suivante :
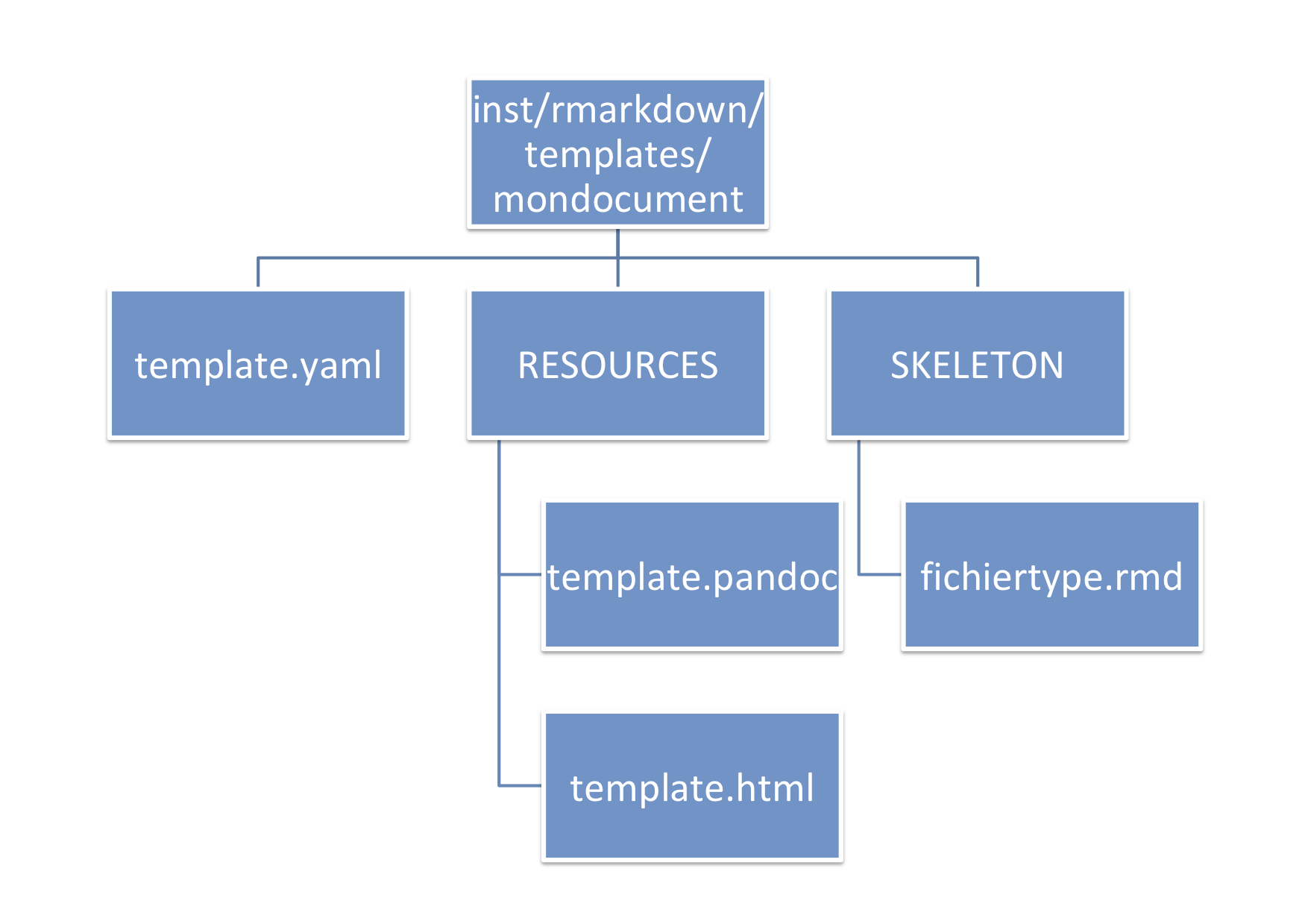
arborescence
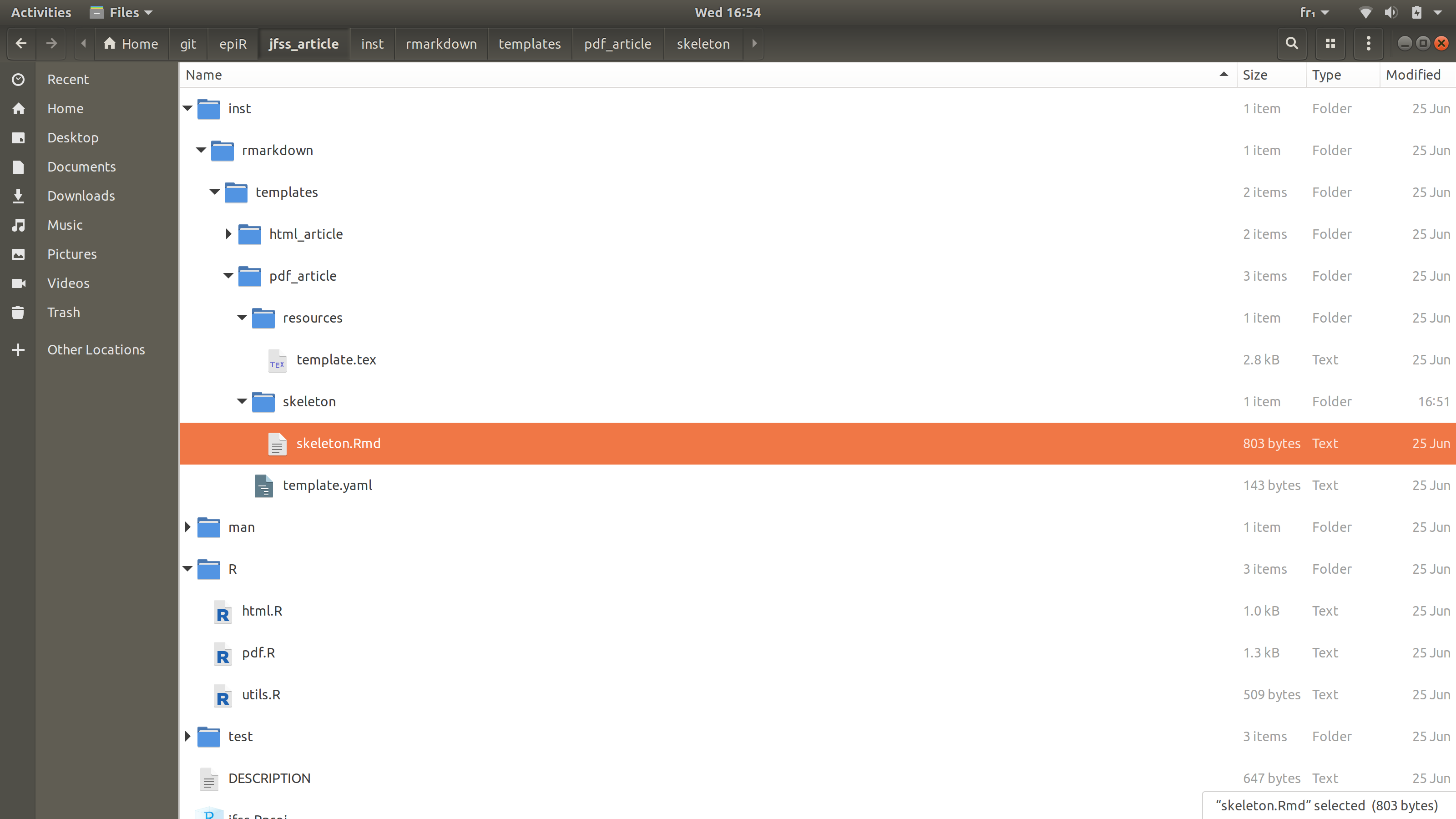
arborescence type
Définition d’un processiong de sortie en HTML
Le fichier html.R contient la fonction html_article() assurant la chaîne de traîtement. Ici, un exemple simple se basant sur la fonction existante html_document()
html_article <- function(...) {
## call the base html_document format with the appropriate options
rmarkdown::html_document(theme = "united",
highlight = "tango",
toc = TRUE,
df_print = "paged",
code_folding = "show",
code_download = TRUE,
toc_float = TRUE,
number_sections = TRUE,
)
}Définition d’un processiong de sortie en PDF
Le fichier pdf.R contient la fonction pdf_article() assurant la chaîne de traîtement. Ici, un exemple simple se basant sur la fonction existante pdf_document() et complétant légèrement le processus
pdf_article <- function(...) {
## call the base pdf_document format with the appropriate options
format <- rmarkdown::pdf_document(fig_width = 8, fig_height = 5, fig_crop = TRUE,
dev = 'pdf', highlight = "pygments",
citation_package = "natbib",
template = template_resources("pdf_article", "template.tex"), ...)
## create knitr options (ensure opts and hooks are non-null)
knitr_options <- rmarkdown::knitr_options_pdf(8, 5, TRUE, 'pdf')
if (is.null(knitr_options$opts_knit)) knitr_options$opts_knit = list()
if (is.null(knitr_options$knit_hooks)) knitr_options$knit_hooks = list()
## set options
knitr_options$opts_chunk$tidy = TRUE
knitr_options$opts_knit$width = 45
## set hooks for special plot output
knitr_options$knit_hooks$plot <- function(x, options) {
knitr::hook_plot_tex(x, options)
}
## override the knitr settings of the base format and return the format
format$knitr <- knitr_options
format$inherits <- 'pdf_document'
format
}template PANDOC
Le fichier template.tex (resp. l’équivalent style.css) contient le template pandoc pour la sortie tex (respectivement, HTML)
\documentclass[$if(fontsize)$$fontsize$,$endif$$if(lang)$$lang$,$endif$$if(papersize)$$papersize$,$endif$$for(classoption)$$classoption$$sep$,$endfor$]{$documentclass$}
% ams
\usepackage{amssymb,amsmath}
\usepackage{ifxetex,ifluatex}
\usepackage{fixltx2e}
\usepackage[OT1]{fontenc}
\usepackage[utf8]{inputenc}
\usepackage{eurosym}
% graphix
\usepackage{graphicx}
% url
\usepackage{url}
% hyperref
\usepackage{hyperref}
% units.
\usepackage{units}
$if(lang)$
\ifnum 0\ifxetex 1\fi\ifluatex 1\fi=0 % if pdftex
\usepackage[shorthands=off,$for(babel-otherlangs)$$babel-otherlangs$,$endfor$main=$babel-lang$]{babel}
$if(babel-newcommands)$
$babel-newcommands$
$endif$
\else
\usepackage{polyglossia}
\setmainlanguage[$polyglossia-lang.options$]{$polyglossia-lang.name$}
$for(polyglossia-otherlangs)$
\setotherlanguage[$polyglossia-otherlangs.options$]{$polyglossia-otherlangs.name$}
$endfor$
\fi
$endif$
$if(numbersections)$
\setcounter{secnumdepth}{2}
$else$
\setcounter{secnumdepth}{-1}
$endif$
% citations
$if(natbib)$
\usepackage{natbib}
\bibliographystyle{$if(biblio-style)$$biblio-style$$else$plainnat$endif$}
$endif$
$if(biblatex)$
\usepackage{biblatex}
$for(bibliography)$
\addbibresource{$bibliography$}
$endfor$
$endif$
% pandoc syntax highlighting
$if(highlighting-macros)$
$highlighting-macros$
$endif$
% multiplecol
\usepackage{multicol}
% strikeout
\usepackage[normalem]{ulem}
% tightlist macro required by pandoc >= 1.14
\providecommand{\tightlist}{%
\setlength{\itemsep}{0pt}\setlength{\parskip}{0pt}}
% title / author / date
$if(title)$
\title{$title$}
$endif$
$if(author)$
\author{$for(author)$$author$$sep$ \and $endfor$}
$endif$
$if(date)$
\date{$date$}
$else$
\date{}
$endif$
%% colored links, setting 'borrowed' from RJournal.sty with 'Thanks, Achim!'
\RequirePackage{color}
\definecolor{link}{rgb}{0.1,0.1,0.8} %% blue with some grey
\hypersetup{
colorlinks,%
citecolor=link,%
filecolor=link,%
linkcolor=link,%
urlcolor=link
}
$for(header-includes)$
$header-includes$
$endfor$
\begin{document}
$if(title)$
\maketitle
$endif$
$if(abstract)$
\begin{abstract}
\noindent $abstract$
\end{abstract}
$endif$
$for(include-before)$
$include-before$
$endfor$
$if(toc)$
{
$if(colorlinks)$
\hypersetup{linkcolor=$if(toccolor)$$toccolor$$else$black$endif$}
$endif$
\setcounter{tocdepth}{$toc-depth$}
\tableofcontents
}
$endif$
$if(lot)$
\listoftables
$endif$
$if(lof)$
\listoffigures
$endif$
$body$
$if(natbib)$
$if(bibliography)$
$if(biblio-title)$
$if(book-class)$
\renewcommand\bibname{$biblio-title$}
$else$
\renewcommand\refname{$biblio-title$}
$endif$
$endif$
\bibliography{$for(bibliography)$$bibliography$$sep$,$endfor$}
$endif$
$endif$
$if(biblatex)$
\printbibliography$if(biblio-title)$[title=$biblio-title$]$endif$
$endif$
$for(include-after)$
$include-after$
$endfor$
\end{document}Définition d’un squelette pour l’utilisateur
Le fichier skeleton.Rmd contient le squelette du fichier Rmd auquel on accède via l’interface Rstudio lors de la création d’un nouveau document, dont les spécification sont définies dans teamplate.yml
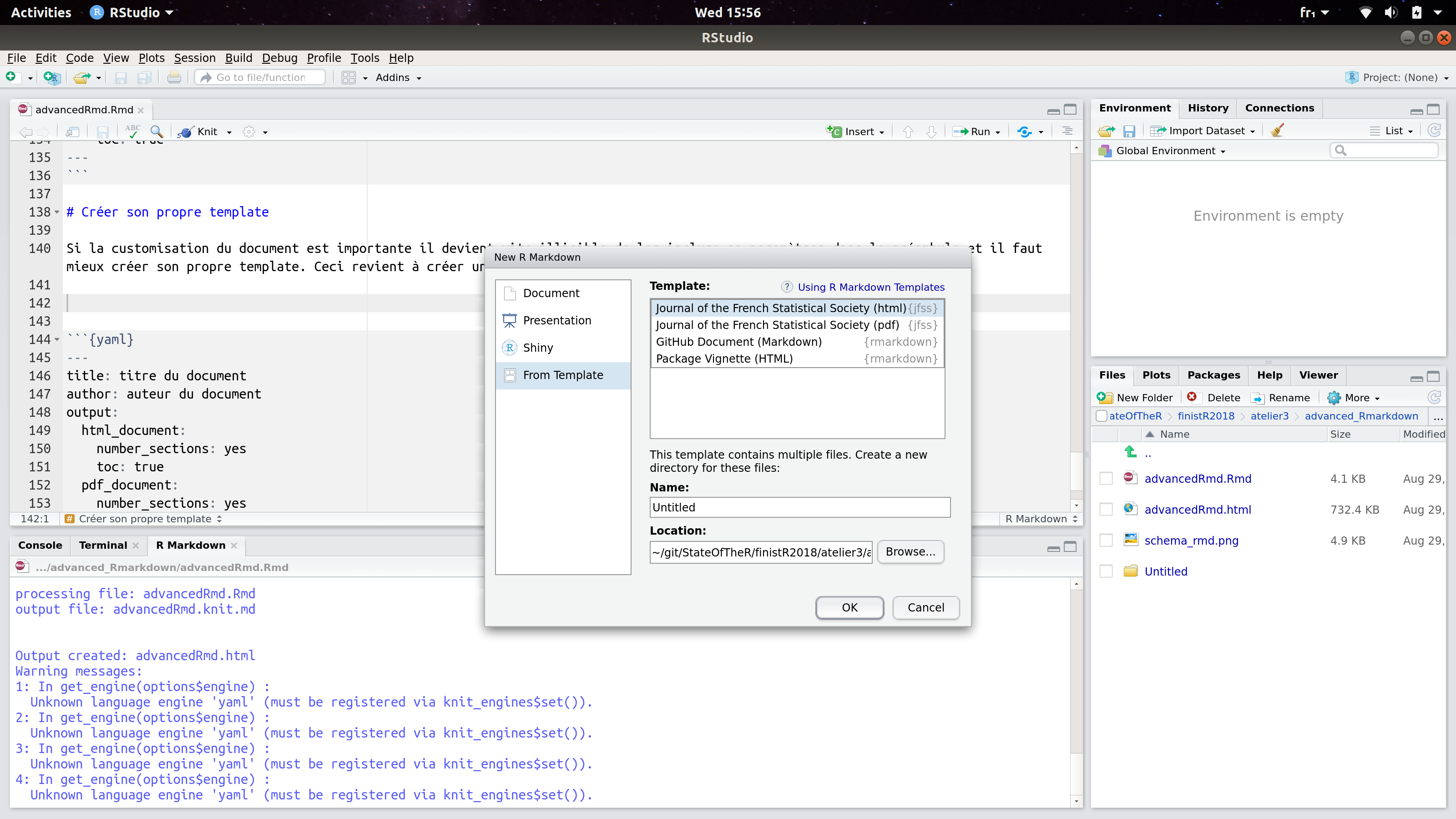
création Rmd via Rstudio