lm en python
FinistR
26/08/2021
Objectif
Le but de ce tutoriel est de faire un TP de modèle linéaire en python, en retrouvant tout les sorties de la fonction lm usuelle
Python depuis rstudio
Package reticulate
On peut appeler python depuis Rstudio grâce au package reticulate.
library(reticulate)Après avoir chargé ce package, toute instruction en python sera reconnue comme tel dans la console!
Installation des librairies python depuis R
Pour installer les librairies, on utilisera les instructions R suivantes:
# Run only once
install_miniconda() ## pour installer miniconda## [1] "/github/home/.local/share/r-miniconda"py_install("pandas") # Pour la manipulation de données
py_install("numpy") # Librairie scientifique
py_install("scipy") # Librairie scientifique
py_install("scikit-learn") # Librairie de machine learning
py_install("seaborn") # Librairie graphique
py_install("statsmodels") # Librairie de modèles statistiques
py_install("yellowbrick") # Librairie graphiqueImportation des modules python
Ceci étant fait, on peut ne plus faire que du python!
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import sklearn as sk
import numpy as np
import statsmodels.api as sm
import statsmodels.formula.api as smf
import scipy.stats as stats
from yellowbrick.datasets import load_concrete
from yellowbrick.regressor import ResidualsPlot
from yellowbrick.regressor import CooksDistance
from sklearn.linear_model import LinearRegression
from sklearn import datasets
plt.style.use('ggplot') # if you are an R user and want to feel at home
from statsmodels.graphics.gofplots import ProbPlotImportation, manipulation et description des données
On utilisera la library de python pandas pour manipuler les données
Import
Ensuite, on importe la librairie, et on charge les données, à la manière de read.table.
import pandas
bats = pandas.read_csv("bats.csv", # Nom du fichier
sep = ";", # Separateur de champs
skiprows = 3, # On ignore les 3 premieres lignes
header = 0) # La premiere ligne correspond au nom de colonnes
print(bats) ## Species Diet Clade ... AUD MOB HIP
## 0 Rousettus aegyptiacus 1 I ... 9.88 105.77 125.97
## 1 Epomops franqueti 1 I ... 10.44 107.80 159.80
## 2 Eonycteris spelaea 1 I ... 5.48 67.00 97.70
## 3 Cynopterus sphinx 1 I ... 4.77 65.27 95.40
## 4 Dobsonia praedatrix 1 I ... 7.09 213.43 233.30
## .. ... ... ... ... ... ... ...
## 58 Coleura afra 3 IV ... 4.08 3.98 12.40
## 59 Taphozous saccolaimus 3 IV ... 9.65 10.92 26.38
## 60 Kerivoula papilosa 3 IV ... 4.47 2.52 19.02
## 61 Myotis myotis 3 IV ... 7.50 5.23 16.24
## 62 Miniopterus medius 3 IV ... 4.77 5.31 17.28
##
## [63 rows x 8 columns]Description
# En-tete des donnees
bats.head()
# Dimension## Species Diet Clade BOW BRW AUD MOB HIP
## 0 Rousettus aegyptiacus 1 I 136.3 2070.00 9.88 105.77 125.97
## 1 Epomops franqueti 1 I 120.0 2210.00 10.44 107.80 159.80
## 2 Eonycteris spelaea 1 I 58.7 1310.00 5.48 67.00 97.70
## 3 Cynopterus sphinx 1 I 48.3 1184.33 4.77 65.27 95.40
## 4 Dobsonia praedatrix 1 I 184.0 3028.00 7.09 213.43 233.30bats.shape
# Type des colonnes## (63, 8)bats.dtypes## Species object
## Diet int64
## Clade object
## BOW float64
## BRW float64
## AUD float64
## MOB float64
## HIP float64
## dtype: objectbats.info()## <class 'pandas.core.frame.DataFrame'>
## RangeIndex: 63 entries, 0 to 62
## Data columns (total 8 columns):
## # Column Non-Null Count Dtype
## --- ------ -------------- -----
## 0 Species 63 non-null object
## 1 Diet 63 non-null int64
## 2 Clade 63 non-null object
## 3 BOW 63 non-null float64
## 4 BRW 63 non-null float64
## 5 AUD 63 non-null float64
## 6 MOB 63 non-null float64
## 7 HIP 63 non-null float64
## dtypes: float64(5), int64(1), object(2)
## memory usage: 4.1+ KBChangement de type d’une colonne
Description d’une colonne facteur du data.frame:
bats["Diet"] = bats["Diet"].apply(str) # Transformation de float a string
bats.Diet.describe() # Quelques statistiques## count 63
## unique 4
## top 1
## freq 29
## Name: Diet, dtype: objectbats["Diet"].value_counts() # Comptage des occurences## 1 29
## 3 27
## 2 5
## 4 2
## Name: Diet, dtype: int64Première figure
plt.figure() # On specifie l'ouverture d'une figure
# Toutes les instructions suivantes se superposerontbats.plot(x ='BOW', y = 'BRW', kind ="scatter",
figsize = [8,6],
color ="b", alpha = 0.3,
fontsize = 14)## <AxesSubplot:xlabel='BOW', ylabel='BRW'>plt.title("BRW vs. BOW",
fontsize = 24, color="darkred")## Text(0.5, 1.0, 'BRW vs. BOW')plt.xlabel("Body weight", fontsize = 18) ## Text(0.5, 0, 'Body weight')plt.ylabel("Brain weight", fontsize = 18)## Text(0, 0.5, 'Brain weight')plt.show() # On montre la figure## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.
## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.
## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.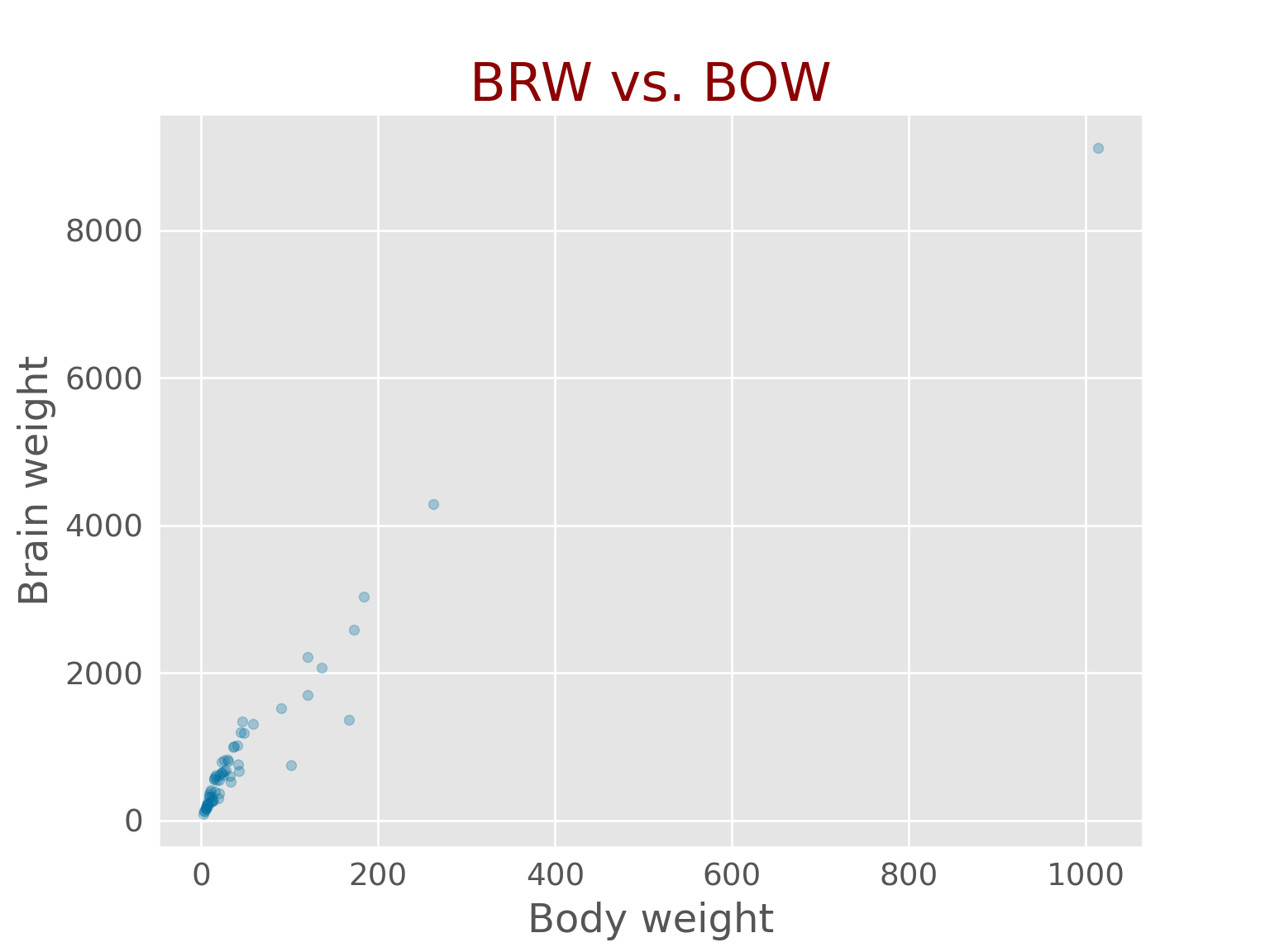
Selection logique
Pour extraire une sous partie des données, on peut utiliser la sélection par un vecteur logique.
# bats.Diet recupere la colonne Diet dans bats
condition_phyto = bats.Diet == '1' # Une colonne de condition
condition_BOW = bats.BOW < 400 # Outlier
batsphyto = bats[condition_phyto & condition_BOW]
batsphyto## Species Diet Clade ... AUD MOB HIP
## 0 Rousettus aegyptiacus 1 I ... 9.88 105.77 125.97
## 1 Epomops franqueti 1 I ... 10.44 107.80 159.80
## 2 Eonycteris spelaea 1 I ... 5.48 67.00 97.70
## 3 Cynopterus sphinx 1 I ... 4.77 65.27 95.40
## 4 Dobsonia praedatrix 1 I ... 7.09 213.43 233.30
## 5 Eidolon helvum 1 I ... 12.77 208.70 258.10
## 7 Macroglossus miniumus 1 I ... 2.40 30.05 52.95
## 8 Syconycteris australis 1 I ... 2.13 31.40 53.10
## 9 Nyctimene albiventer 1 I ... 4.56 68.93 81.40
## 23 Brachyphylla cavernarum 1 II ... 8.63 42.20 78.80
## 24 Lionycteris spurrelli 1 II ... 3.71 10.30 29.50
## 25 Glossophaga soricina 1 II ... 3.74 12.20 35.00
## 26 Leptonycteris curasoae 1 II ... 5.57 18.60 44.95
## 27 Anoura geofroyi 1 II ... 5.20 14.15 41.40
## 28 Phylloderma stenops 1 II ... 10.20 87.40 91.70
## 29 Phyllostomus haustatus 1 II ... 12.74 34.33 65.60
## 30 Mimon crenulatum 1 II ... 5.92 7.30 18.20
## 31 Trachops cirrhosus 1 II ... 16.34 23.50 50.60
## 32 Tonatia bidens 1 II ... 13.37 17.96 28.30
## 33 Vampyrum spectrum 1 II ... 27.60 92.00 110.40
## 34 Micronycteris brachyotis 1 II ... 4.19 13.85 17.10
## 35 Carollia perspicillata 1 II ... 5.27 23.55 40.75
## 36 Rhinophlylla pumilio 1 II ... 4.57 18.80 30.30
## 37 Sturnira lilium 1 II ... 4.77 30.77 49.73
## 38 Artibeus lituratus 1 II ... 7.21 34.38 54.90
## 39 Uroderma bilobatum 1 II ... 5.98 28.70 42.70
## 40 Vampyrops vittatus 1 II ... 11.56 29.22 52.46
## 41 Chiroderma villosum 1 II ... 7.95 28.75 47.58
##
## [28 rows x 8 columns]Régression linéaire
Avec scikit-learn
Pour ajuster une regression linéaire, on peut utiliser la librairie scikit-learn, librairie de machine learning:
import numpy as np
import sklearn.linear_model as sklm
# Les features ("X") doivent être données sous forme de matrice
X = np.asarray(batsphyto.BOW).reshape(-1, 1)
regression_simple = sklm.LinearRegression()
regression_simple.fit(X = X, y = batsphyto.BRW)
# Coefficients de la régression## LinearRegression()beta0, beta1 = [regression_simple.intercept_, regression_simple.coef_]
beta0, beta1## (346.5451556610758, array([14.50990083]))Cette librairie ne donnera quasiment aucun diagnostic statistique usuel (test sur les paramètres, intervalles de confiance, etc..)
Avec statsmodels
Pour une approche plus statistique, et proche de celle de R, on utilisera statsmodels, dont la documentation est très détaillée
import statsmodels.api as sm
import statsmodels.formula.api as smf
from statsmodels.sandbox.regression.predstd import wls_prediction_std
import matplotlib.pyplot as pltLa syntaxe est très proche de R!
regression = smf.ols("BRW ~ BOW", data = batsphyto).fit()
print(regression.summary())## OLS Regression Results
## ==============================================================================
## Dep. Variable: BRW R-squared: 0.978
## Model: OLS Adj. R-squared: 0.977
## Method: Least Squares F-statistic: 1147.
## Date: Thu, 02 Sep 2021 Prob (F-statistic): 4.91e-23
## Time: 10:48:11 Log-Likelihood: -177.41
## No. Observations: 28 AIC: 358.8
## Df Residuals: 26 BIC: 361.5
## Df Model: 1
## Covariance Type: nonrobust
## ==============================================================================
## coef std err t P>|t| [0.025 0.975]
## ------------------------------------------------------------------------------
## Intercept 346.5452 35.492 9.764 0.000 273.590 419.500
## BOW 14.5099 0.429 33.860 0.000 13.629 15.391
## ==============================================================================
## Omnibus: 0.012 Durbin-Watson: 1.654
## Prob(Omnibus): 0.994 Jarque-Bera (JB): 0.099
## Skew: 0.016 Prob(JB): 0.952
## Kurtosis: 2.711 Cond. No. 110.
## ==============================================================================
##
## Notes:
## [1] Standard Errors assume that the covariance matrix of the errors is correctly specified.On peut representer graphiquement la droite de regression ainsi que les intervalles de confiance associés
# Calcul
fig = sm.graphics.plot_fit(regression, "BOW")
plt.show()## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.
## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.
## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.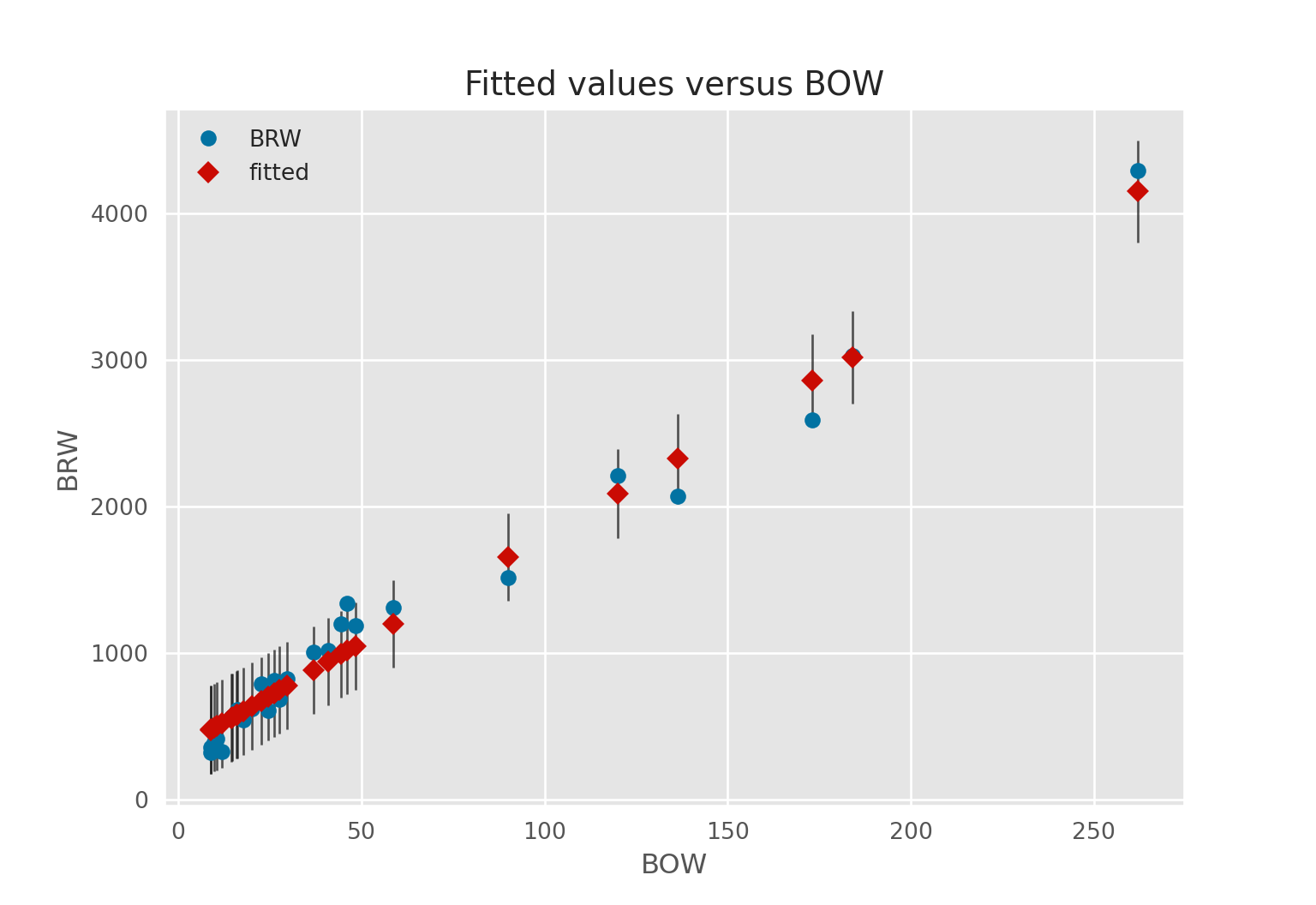
Tests sur les paramètres
On peut tester si une combinaison linéaire des paramètres est égale à 0:
combinaisons_lineaires = "Intercept = 0, BOW = 0, BOW - Intercept = 0, 3 * Intercept = 2 * BOW"
print(regression.t_test(combinaisons_lineaires))## Test for Constraints
## ==============================================================================
## coef std err t P>|t| [0.025 0.975]
## ------------------------------------------------------------------------------
## c0 346.5452 35.492 9.764 0.000 273.590 419.500
## c1 14.5099 0.429 33.860 0.000 13.629 15.391
## c2 -332.0353 35.775 -9.281 0.000 -405.571 -258.500
## c3 1010.6157 107.040 9.441 0.000 790.592 1230.640
## ==============================================================================Tests des effets
On peut tester les effets
from statsmodels.stats.anova import anova_lm
regression_multiple = smf.ols('BRW ~ BOW + AUD', data = batsphyto).fit()
anovaResults = anova_lm(regression_multiple, typ = "II") # On peut choisir le type
print(anovaResults)## sum_sq df F PR(>F)
## BOW 1.623042e+07 1.0 788.051516 2.007592e-20
## AUD 7.599175e+03 1.0 0.368970 5.490443e-01
## Residual 5.148909e+05 25.0 NaN NaNTests de modèles emboités
anovaResults = anova_lm(regression, regression_multiple) # On peut choisir le type
print(anovaResults)## df_resid ssr df_diff ss_diff F Pr(>F)
## 0 26.0 522490.111038 0.0 NaN NaN NaN
## 1 25.0 514890.935969 1.0 7599.175069 0.36897 0.549044ANCOVA
Il semblerait que l’inclusion des variables qualitatives se déroule de la même manière que dans R. Exemple sur l’ANCOVA:
ancova = smf.ols('BRW ~ Clade * BOW', data = batsphyto).fit()
print(ancova.summary())## OLS Regression Results
## ==============================================================================
## Dep. Variable: BRW R-squared: 0.979
## Model: OLS Adj. R-squared: 0.977
## Method: Least Squares F-statistic: 380.3
## Date: Thu, 02 Sep 2021 Prob (F-statistic): 2.33e-20
## Time: 10:48:17 Log-Likelihood: -176.38
## No. Observations: 28 AIC: 360.8
## Df Residuals: 24 BIC: 366.1
## Df Model: 3
## Covariance Type: nonrobust
## ===================================================================================
## coef std err t P>|t| [0.025 0.975]
## -----------------------------------------------------------------------------------
## Intercept 379.6310 73.819 5.143 0.000 227.276 531.986
## Clade[T.II] -21.6150 85.999 -0.251 0.804 -199.108 155.878
## BOW 14.5476 0.587 24.803 0.000 13.337 15.758
## Clade[T.II]:BOW -0.8777 1.045 -0.840 0.409 -3.034 1.278
## ==============================================================================
## Omnibus: 1.791 Durbin-Watson: 1.808
## Prob(Omnibus): 0.408 Jarque-Bera (JB): 0.733
## Skew: 0.324 Prob(JB): 0.693
## Kurtosis: 3.457 Cond. No. 350.
## ==============================================================================
##
## Notes:
## [1] Standard Errors assume that the covariance matrix of the errors is correctly specified.Moyennes ajustées
Il semblerait que, pour le moment, le calcul des moyennes ajustées doivent se faire à la main!
Graphes de diagnostics
On accède aux prédictions ou aux résidus avec les différents attributs de l’objet regression.
# model predictions
model_fitted_y = regression.fittedvalues
# model residuals
model_residuals = regression.residIl existe différents graphes de diagnostics qui diffèrent un peu de ceux de R
fig = plt.figure(figsize=(15,8))
sm.graphics.plot_regress_exog(regression, 'BOW', fig = fig)plt.show()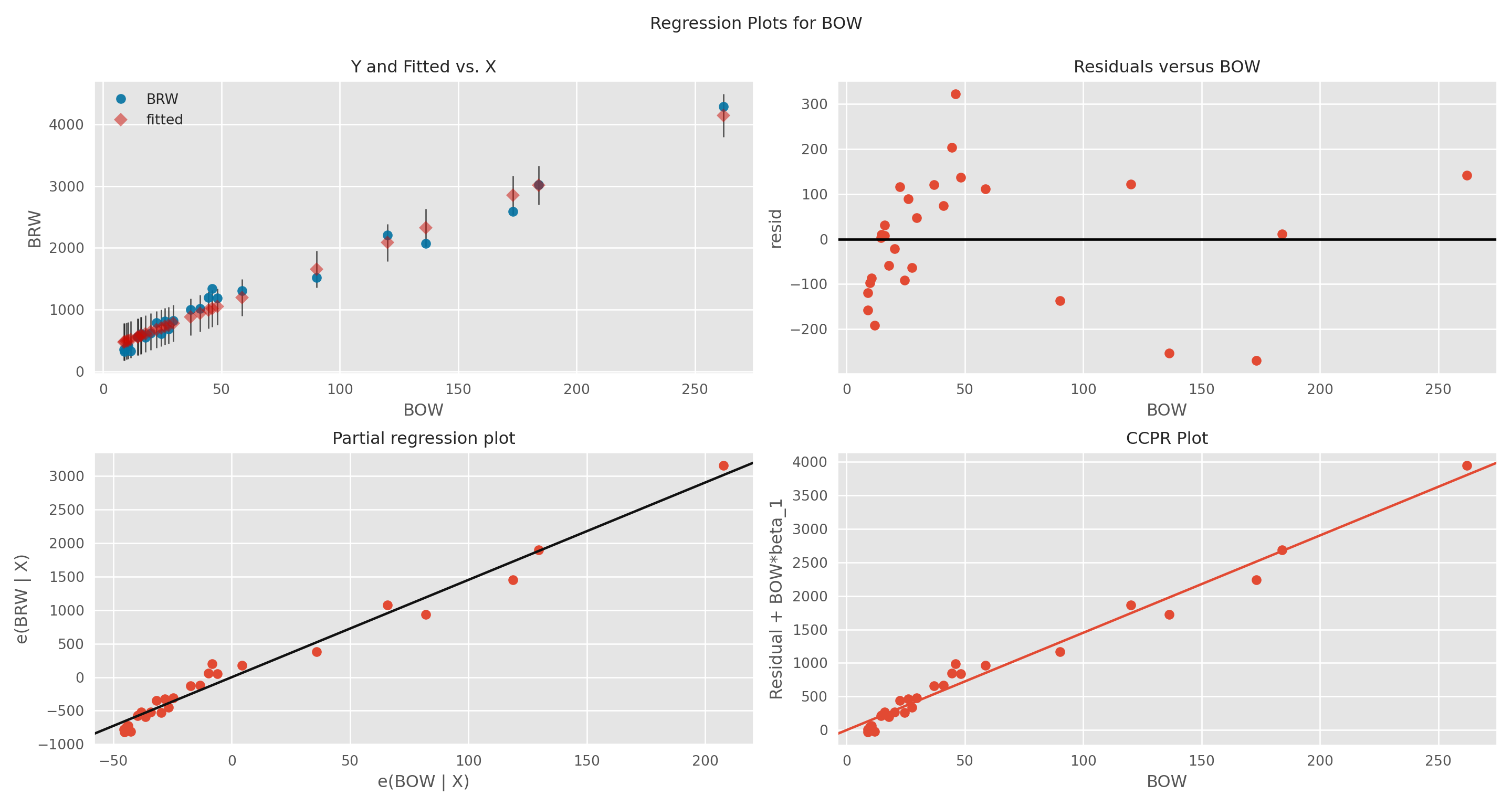
Graphique de l’influence
On peut obtenir le vecteur des distances de Cook grâce à la méthode get_influence
distances_cook = regression.get_influence().cooks_distance[0]On peut tracer résidus standardisés en fonction du levier grâce à une fonction dédiée, graphique sans légende…
fig = sm.graphics.influence_plot(regression, criterion="cooks", alpha = 0.005)
plt.show()## findfont: Font family ['sans-serif'] not found. Falling back to DejaVu Sans.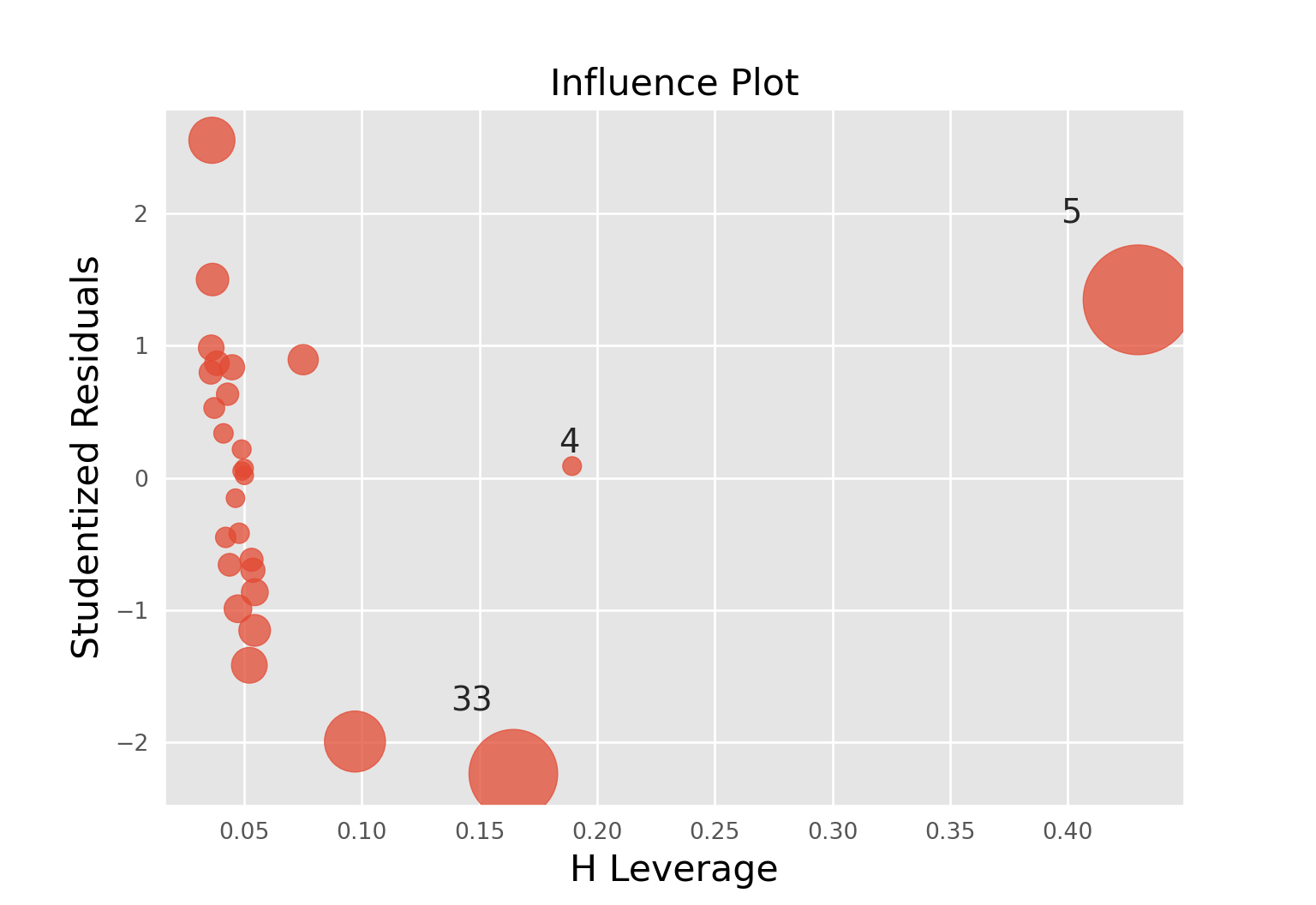
plt.figure(figsize=(6,5))fig = sm.qqplot(regression.resid, stats.t, distargs=(4,), fit=True, line="45")
plt.show()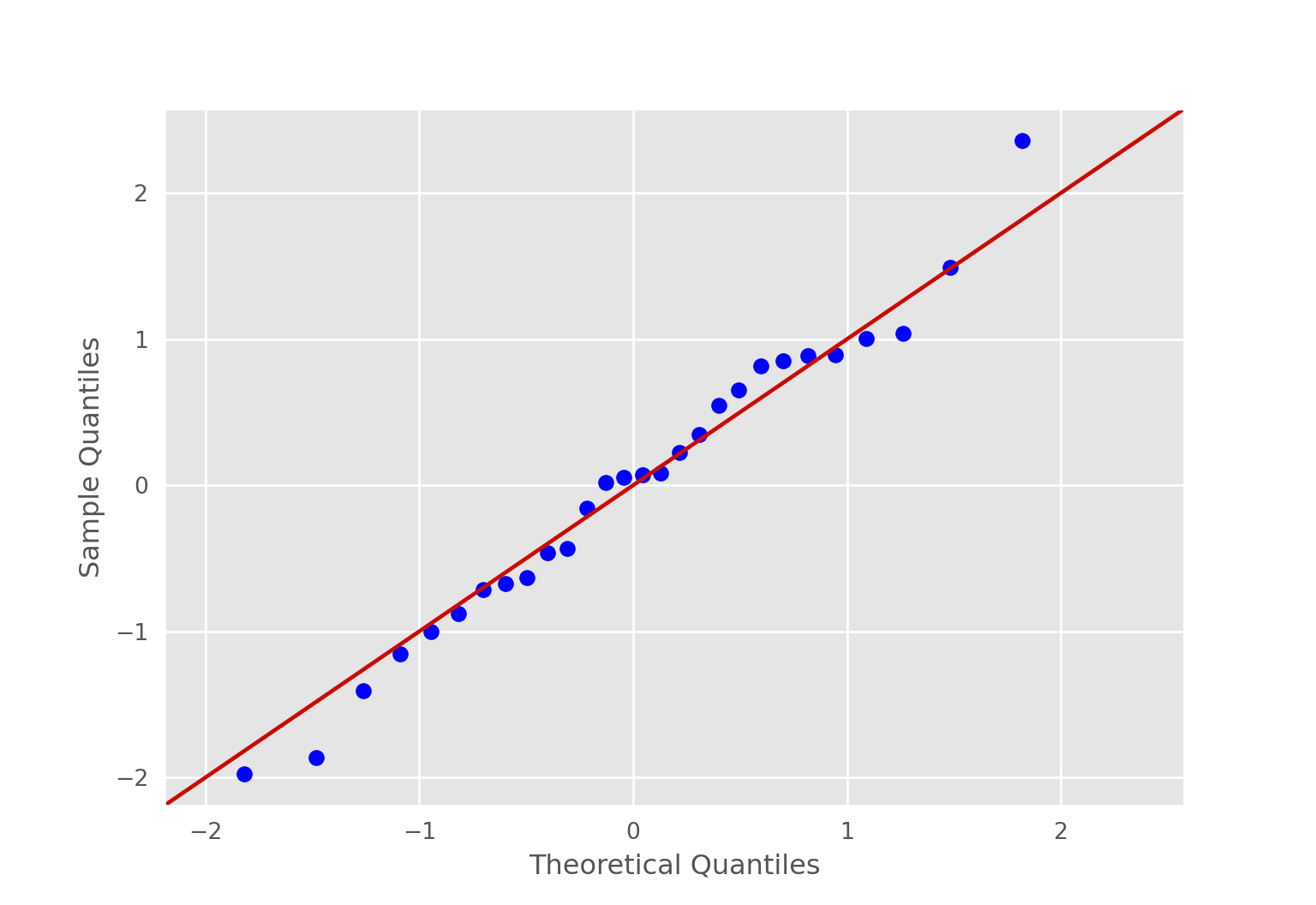
Quelques graphiques avec seaborn
Corrélations
# correlation heatmap
bats3=bats[["BRW","BOW","AUD","MOB","HIP"]]
corr = bats3.corr()
plt.figure(figsize = (6,6))sns.heatmap(corr, cmap="RdBu",
xticklabels=corr.columns.values,
yticklabels=corr.columns.values)## <AxesSubplot:>plt.show()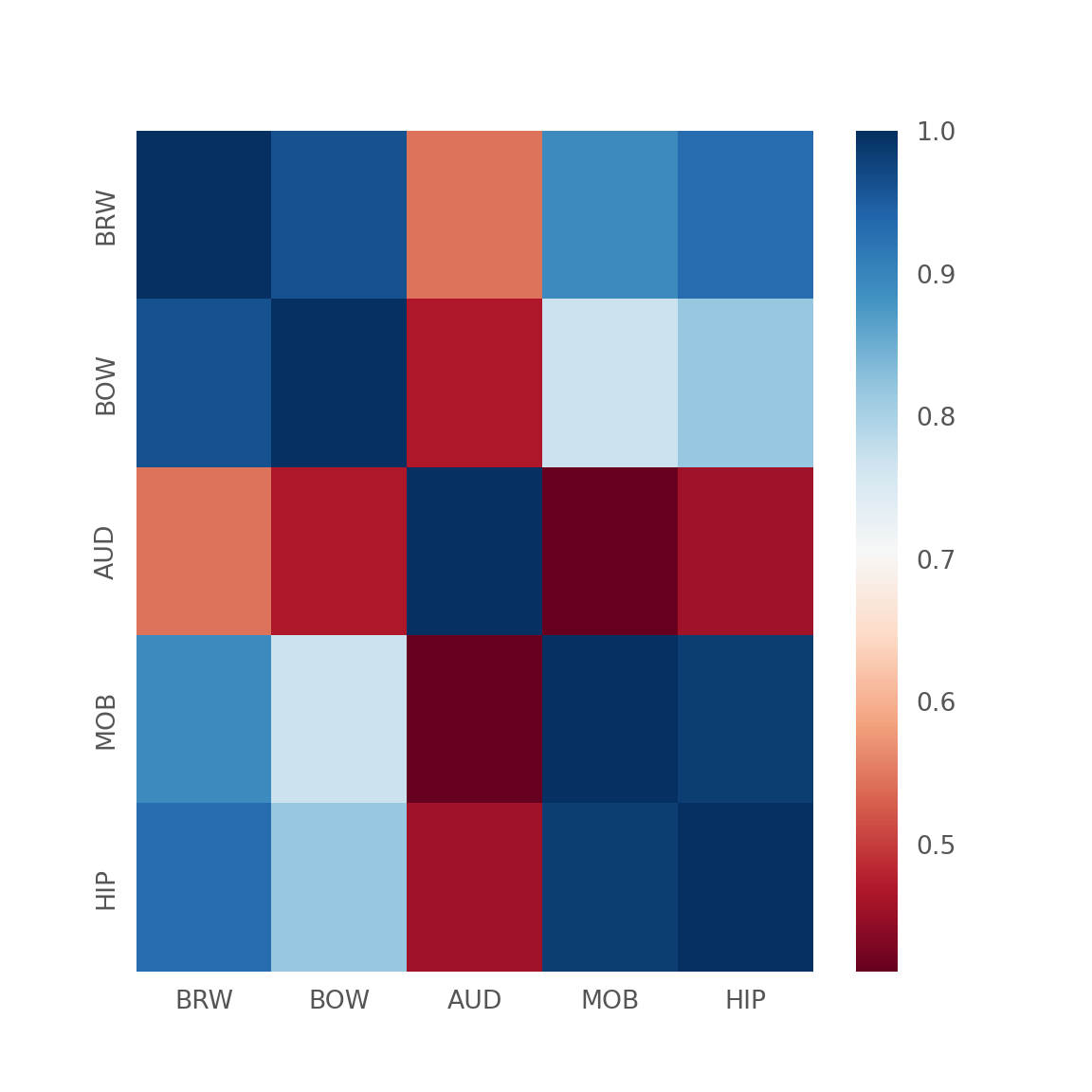
Boites à moustache
Attention, la variable groupe doit être en caractère impérativement.
sns.boxplot(x="BOW", y="Clade", data=bats,
width=.6, palette="vlag")
# Add in points to show each observation## <AxesSubplot:xlabel='BOW', ylabel='Clade'>sns.stripplot(x="BOW", y="Clade", data=bats,
size=4, color=".3", linewidth=0)## <AxesSubplot:xlabel='BOW', ylabel='Clade'>plt.show()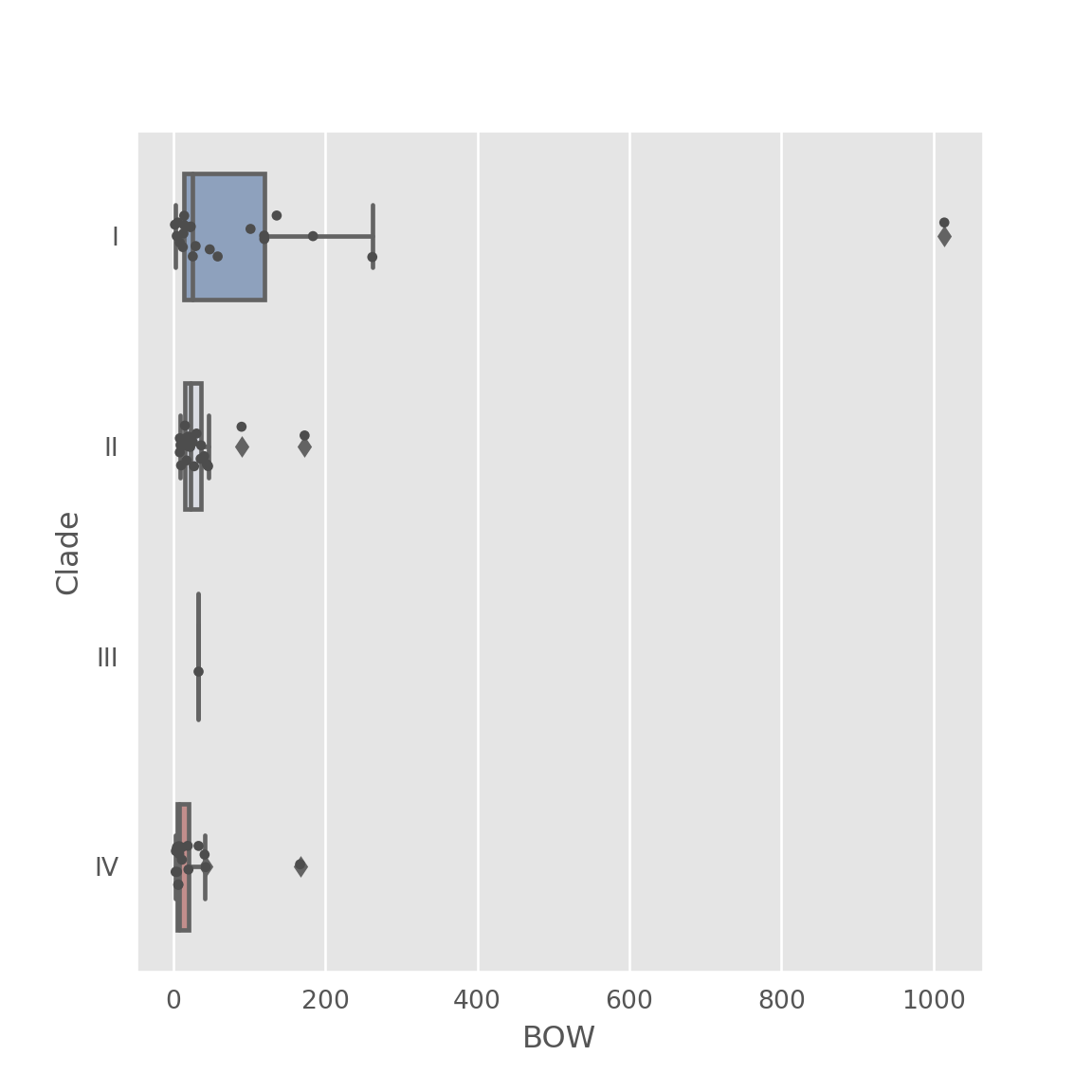
Pair plot
sns.set(style="ticks", color_codes=True)
sns.pairplot(bats3)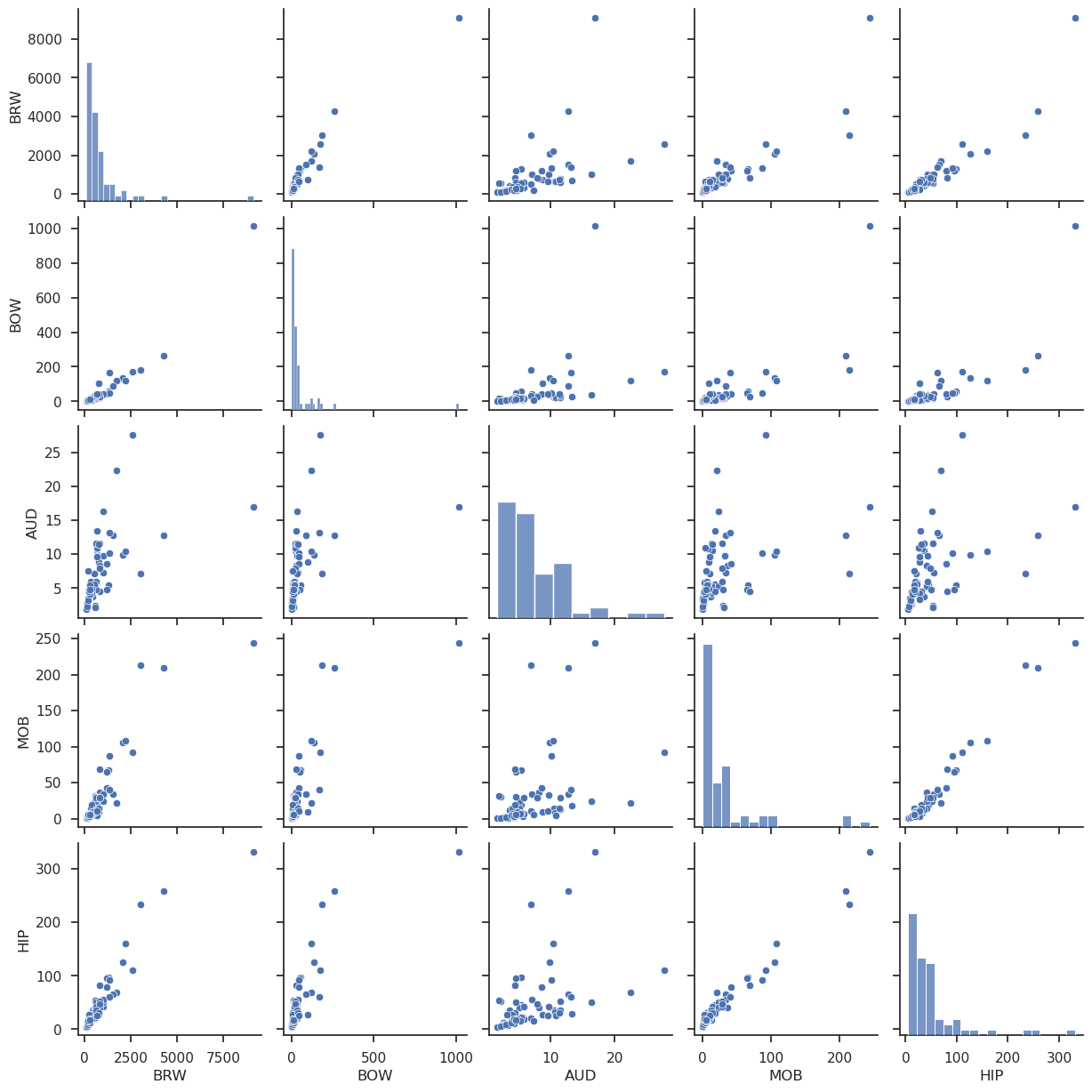
sns.pairplot(bats, hue="Clade")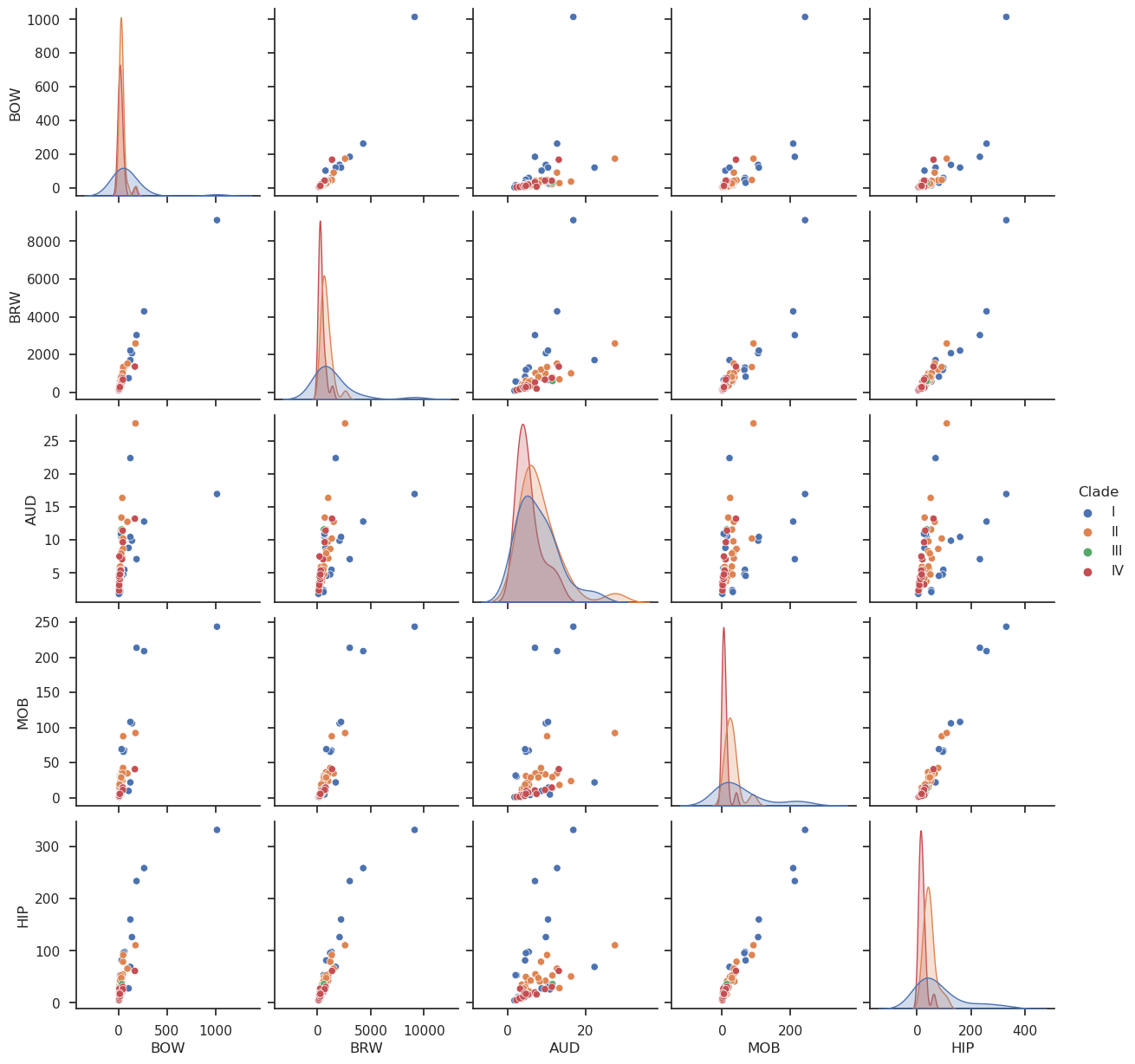
Regression plot
Attention les intervalles de confiance sont faits par bootstrap!
plt.figure(figsize = (5,5))sns.regplot(x="BOW", y="BRW", data=bats);
plt.show()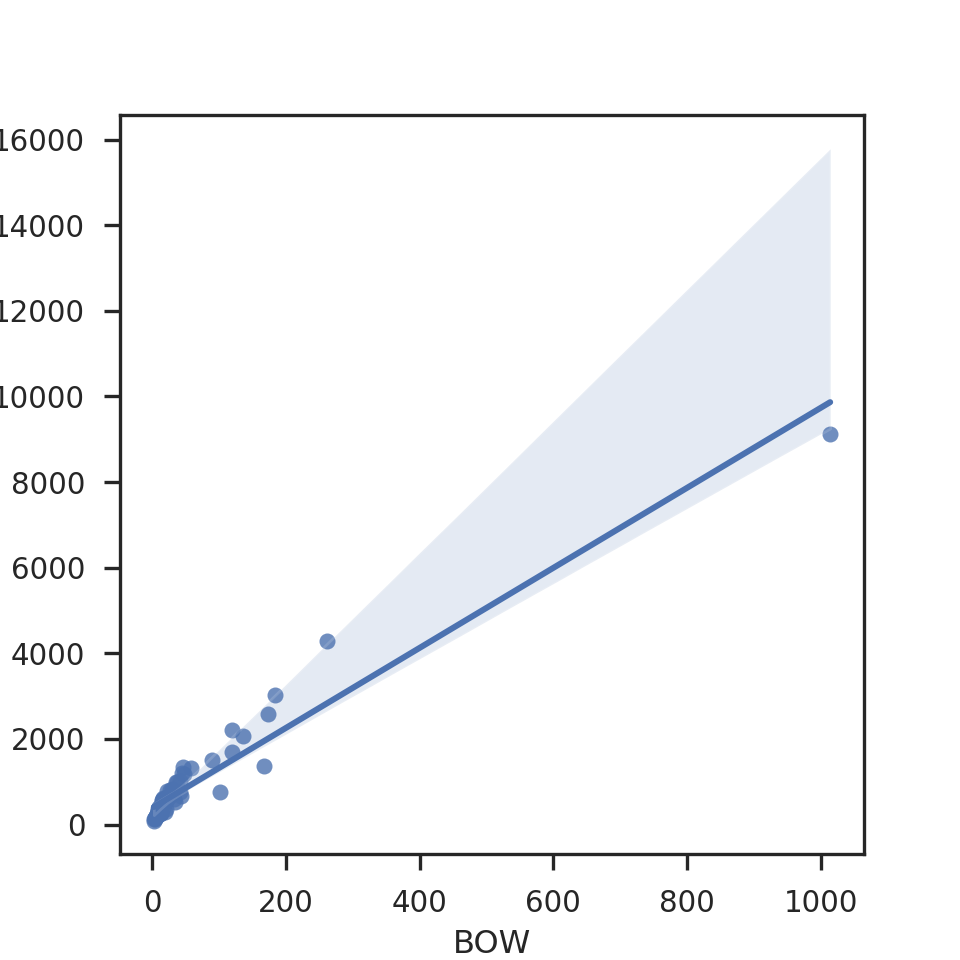
# le ; est indispensable pour que le graph s'affiche!
sns.lmplot(x="BOW", y="BRW", hue="Diet", data=bats);
plt.show()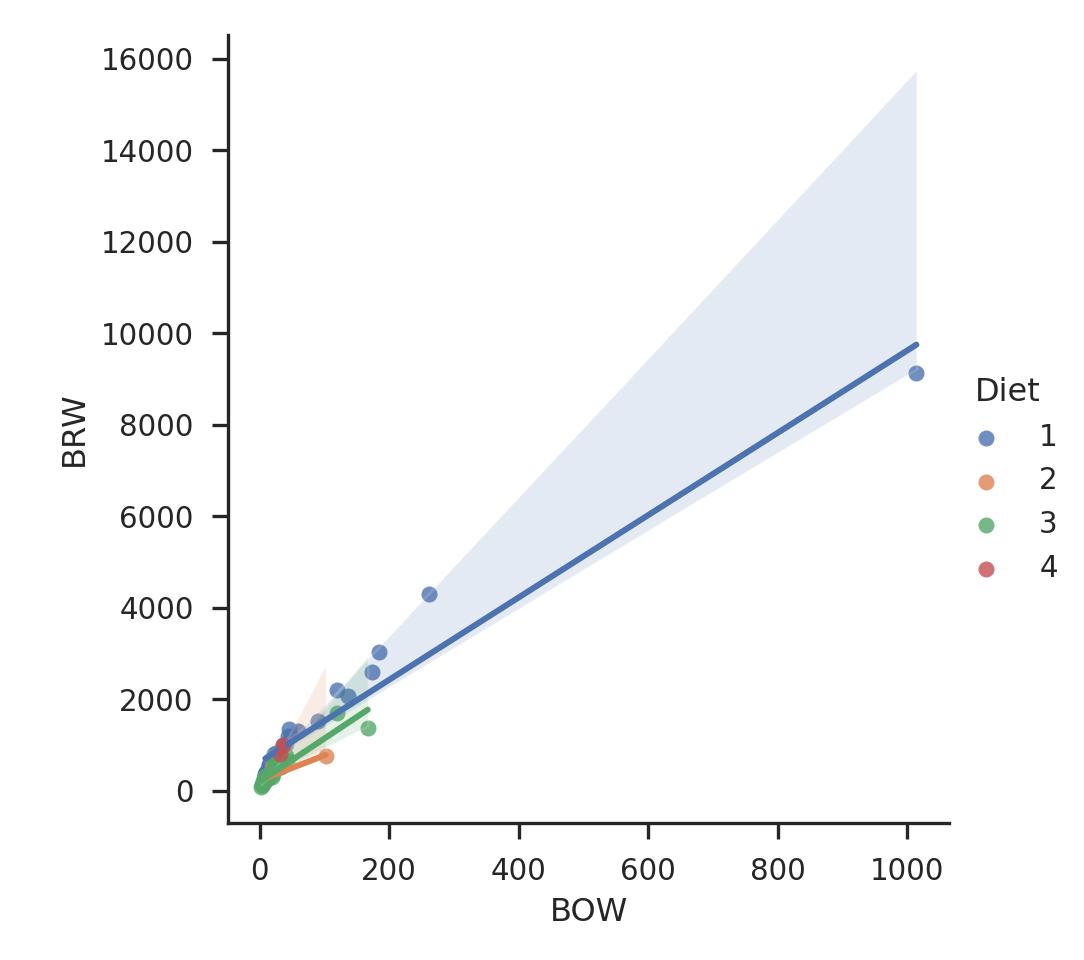
Complément sur les packages Python pour faire vos graphiques
- bqplot : pour les graphes interactifs intégrés dans des notebooks.
- seaborn : destinés aux statistiques et construit sur matplotlib.
- toyplot : excellent pour tracer les graphes avec un très beau rendu pour le web.
- bokeh : interactif qui fonctionne en mode serveur, c’est le Rshiny de Python.
- pygal : si vous aimez les histogrammes et les camemberts.
- Altair : pour tracer à partir d’une dataframe, construit sur la bibliothèque javascript vega. Magnifique rendu en html.
- plot.ly : interactif et orienté sciences des données, fonctionne aussi avec R et Julia.
- YT: orienté plus pour la physique mais excellent pour tracer des contours, des volumes, des surfaces et des particules.
- Yellowbrick : compagnon graphique de scikit-learn.
- scikit-plot : utile pour tracer les métriques issues de scikit-learn. Le développement semble à l’arrêt cependant.